Une structure en "épingle à cheveux" contrôle la traduction du génome du virus SARS-CoV-2
Les scientifiques ont démontré que la structure en épingle à cheveux nommée SL1 située dans l’une des extrémités de l’ARN génomique du virus SARS-CoV-2, est indispensable pour détourner le système de traduction de l'hôte vers la synthèse des protéines virales. Ces résultats sont publiés dans la revue RNA.
Les chercheurs ont étudié les mécanismes moléculaires qui permettent la traduction en protéines du génome viral par le ribosome de la cellule infectée. Ce dernier est un gros complexe ribonucléoprotéique sur lequel les ARN messagers, passant au sein d'un étroit "canal", vont pouvoir être traduit en protéines. Lors des étapes précoces de l’infection par le virus, la protéine NSP1 est l’une des premières protéines virales produites. Cette protéine NSP1 va se fixer immédiatement sur le ribosome de la cellule hôte et bloquer le site d’entrée du canal pour interdire l’accès des ARN messagers de la cellule qui ne seront donc plus traduits. Le virus prend ainsi le contrôle de la machinerie traductionnelle de la cellule et éteint littéralement la traduction "normale" de la cellule.
Cependant, le génome du virus, lui, continue à être traduit malgré la présence de NSP1 sur le ribosome. Les expériences réalisées par les chercheurs ont permis de comprendre comment le virus peut contourner ce blocage grâce à la protéine NSP1. En effet, le virus possède en amont de chacun de ses ARN messagers, une petite structure "en épingle à cheveux" appelée SL1 qui lui permet d’ouvrir l’accès au ribosome en agissant sur NSP1. Le virus possède en quelque sorte une clé qui lui autorise la traduction de ses protéines en déverrouillant le ribosome bloqué par la protéine virale NSP1 pendant l’infection.
Cette découverte pourrait ouvrir la voie à de nouvelles approches antivirales visant à bloquer le cycle cellulaire du virus. En effet, le rôle primordial de SL1 dans le processus d'infection fait de cet élément du virus une cible de choix pour le développement de nouvelles molécules inhibitrices capables de bloquer spécifiquement l’action de SL1 et permettant d’éteindre la traduction du virus. SL1 pourrait ainsi devenir un véritable ‘talon d’Achille’ du virus.
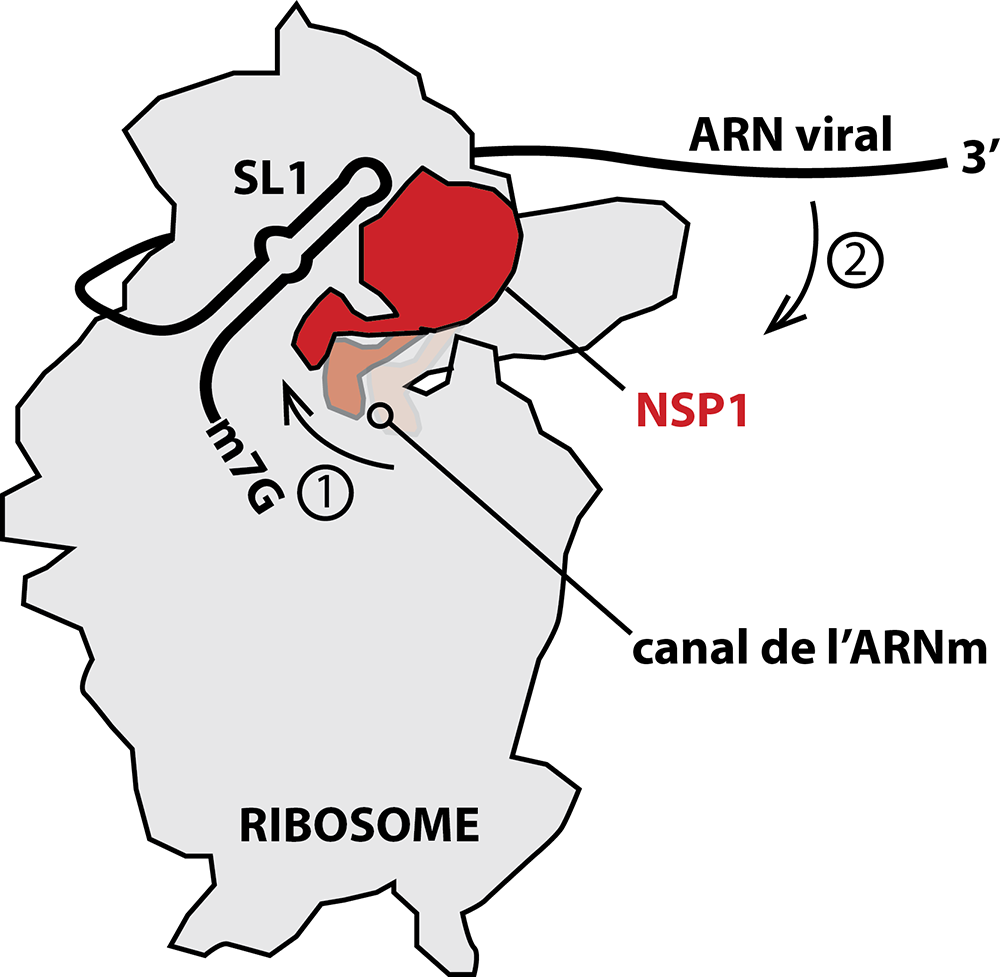
Figure : Lors de l’infection par le SARS-CoV-2, la première protéine virale produite est la protéine NSP1. Cette protéine se fixe sur le ribosome de la cellule infectée et bouche l’entrée du canal des ARN messagers. C’est de cette manière que la protéine NSP1 éteint la traduction de la cellule hôte pendant les phases précoces de l'infection. Cependant, la traduction virale n’est pas impactée par la présence de ce verrou moléculaire qu’est NSP1. Pour échapper au blocage par NSP1, les ARN messagers du virus possèdent une clé moléculaire qui permet d’ouvrir l’accès au ribosome en déplaçant la NSP1. Cette clé est une structure en tige boucle appelée SL1 qui est présente dans tous les ARN messagers
Pour en savoir plus
The viral protein NSP1 acts as a ribosome gatekeeper for shutting down host translation and fostering SARS-CoV-2 translation.
Tidu A, Janvier A, Schaeffer L, Sosnowski P, Kuhn L, Hammann P, Westhof E, Eriani G, Martin F.RNA. 2020 Dec 2:rna.078121.120. doi: 10.1261/rna.078121.120. Online ahead of print
Contact
Laboratoire
Architecture et réactivité de l'ARN (ARN) - CNRS / Université de Strasbourg
Institut de Biologie Moléculaire et Cellulaire (CNRS)
2 allée Konrad Roentgen
67084 Strasbourg cedex