Une première analyse par spectroscopie RMN de la protéase 3CLpro du SARS-CoV-2
Dans cette étude, les scientifiques décrivent la première analyse par spectroscopie de résonance magnétique nucléaire (RMN) de la protéase principale du SARS-Cov-2, une cible thérapeutique prometteuse. Cette approche a notamment permis d’identifier une petite molécule avec des propriétés antivirales. Ces données de RMN sont librement accessibles à la communauté scientifique mondiale et permettront de mieux comprendre les mécanismes de régulation complexes de cette enzyme. Ces travaux sont publiés dans la revue Angewandte Chemie International Edition.
La protéase 3CLpro est une enzyme essentielle du SARS-CoV-2 car elle découpe les polyprotéines précurseurs pp1a et pp1ab en différentes protéines actives qui permettront alors la réplication du virus. La 3CLpro est une cible thérapeutique importante car sa séquence en acides aminés est bien conservée chez les coronavirus apparentés et ne possède pas d’homologue chez l’humain.
Les chercheurs ont réalisé la première étude par spectroscopie de Résonance Magnétique Nucléaire (RMN) de la protéase 3CLpro du SARS-CoV-2. En utilisant des bactéries, il été possible de produire la protéine virale, marquées avec des isotopes (2H, 15N et 13C), en grande quantité. L’utilisation d’un spectromètre RMN à très haut champs (900 MHz), faisant partie de l’infrastructure nationale IR-RMN THC, s’est révélée nécessaire en raison de la complexité du système étudié. Ces données de RMN permettent d’analyser des processus d’interactions moléculaires et d’obtenir des informations structurales et dynamiques en solution et à l’échelle des atomes. Les résultats obtenus permettent de comprendre les mécanismes de régulation complexes de cette enzyme, laquelle doit s’assembler en dimère pour permettre à son site actif, doué d’une plasticité importante, d’adopter la conformation adéquate pour accueillir le substrat et le couper ensuite. Des sites de régulation allostérique, éloignés du site actif, ont également été mis en évidence. La compréhension de ces mécanismes est très importante afin de développer des inhibiteurs de son activité enzymatique.
Dans cette optique, les chercheurs ont réalisé un criblage de fragments moléculaires par RMN contre la protéase 3CLpro. Grâce à la petite taille de ces molécules, il est possible d’explorer efficacement l’espace chimique tout en testant un nombre modéré de molécules. A l’issu du criblage, 38 fragments sur les 960 testés ont été identifiés comme se liant à la 3CLpro. L’analyse des signaux RMN a permis d’identifier 3 sites de liaison différents, deux au sein du site actif et un à l’interface de dimérisation.
Le fragment F01, issu du criblage RMN, s’est révélé capable d’inhiber l’activité enzymatique de la protéase 3CLpro et d’inhiber la réplication du SARS-CoV-2 dans des cultures de cellules infectées. Ce fragment devra désormais être optimisé afin d’augmenter son efficacité. Sa structure chimique pourra être modifiée et complexifiée de manière rationnelle en exploitant la structure cristallographique du complexe 3CLpro:F01 que les chercheurs ont résolue à partir des données collectées au synchrotron SOLEIL. Ce fragment F01 pourrait également être utilisé en combinaison avec un autre fragment se liant lui au niveau de l’interface dimérique.
Ces résultats, fondés sur des expertises complémentaires (biologie structurale, chimie médicinale et virologie moléculaire), permettront de mieux comprendre les interactions complexes entre la plasticité du site actif, la dimérisation et l’activité enzymatique de la 3CLpro et constituent un nouvel outil pour assister le développement d’inhibiteurs qui pourraient devenir des médicaments antiviraux.
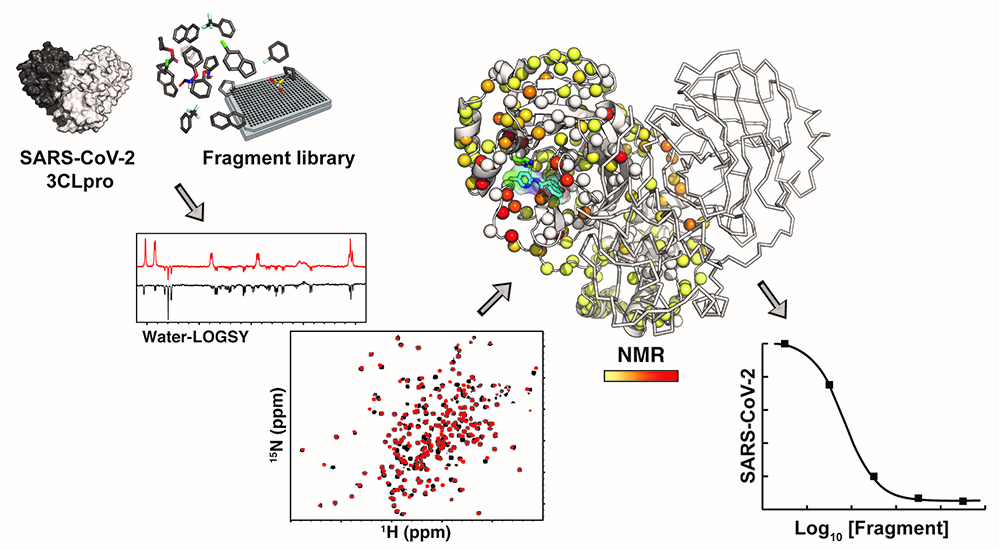
Figure : Une bibliothèque de fragments moléculaires a été criblée contre la protéase 3CLpro du SARS-CoV-2 en utilisant la spectroscopie de résonance magnétique nucléaire (RMN) à haut champ. Le meilleur fragment issu du crible permet d’inhiber le SARS-CoV-2 dans des cellules infectées.
Pour en savoir plus :
NMR spectroscopy of the main protease of SARS-CoV-2 and fragment-based screening identify three protein hotspots and an antiviral fragment.
Cantrelle FX, Boll E, Brier L, Moschidi D, Belouzard S, Landry V, Leroux F, Dewitte F, Landrieu I, Dubuisson J, Deprez B, Charton J, Hanoulle X.
Angew Chem Int Ed Engl. 2021 Sep 27. doi: 10.1002/anie.202109965.
Contact
laboratoire
Biologie Structurale Intégrative (CNRS – INSERM, Institut Pasteur de Lille, Université de Lille, CHU Lille)
1 rue du Professeur Calmette 59019 LILLE CEDEX