Une nouvelle fonction pour une protéine SR : la dégradation des ARNm instables
Il existe des gènes dépourvus d’intron dont le produit est pourtant efficacement exprimé dans la cellule. C’est le cas de la grande majorité des gènes des virus herpes comme le virus d’Epstein-Barr. Ces virus produisent un facteur dont la fonction est de favoriser l’accumulation cytoplasmique des ARN messagers (ARNm). Cette étude publiée le 27 aout 2018 dans la revue Scientific Reports met en évidence le rôle stabilisateur de la protéine virale EB2 sur les ARNm viraux. En interagissant avec la protéine cellulaire SRSF3 impliquée dans l’épissage des ARNm, EB2 empêche le recrutement sur les ARN cibles du complexe RNA-exosome de dégradation des ARNm.
Les virus herpes sont très largement répandus. Neuf virus herpès différents infectent l’homme et sont soit responsables de certaines pathologies, soit associés au développement de certains cancers. Ainsi, par exemple, le virus d’Epstein Barr (EBV) est ubiquitaire. Il est l’agent causal de la mononucléose infectieuse et il est associé au développement de différents cancers touchant soit des cellules lymphoïdes (lymphome de Burkitt, lymphomes post-transplantation) soit des cellules épithéliales (cancer du rhinopharynx, un sous-type de cancer gastrique). Les virus herpes sont des virus à ADN qui se répliquent dans le noyau de leur cellules-hôtes en utilisant différentes machineries cellulaires notamment pour l’expression de leurs gènes.
Chez les eucaryotes, la très grande majorité des gènes contiennent de nombreux introns. La maturation de leurs pré-ARNm est un processus complexe qui génère des ARNm matures via divers mécanismes dont l’épissage, par le spliceosome, des régions introniques transcrites à partir de ces gènes morcelés. Lors de ce processus de maturation, les ARN reconnus comme « anormaux », notamment les ARNm non épissés sont efficacement détruits par les complexes multi-protéiques de type RNA-exosome, initialement décrits chez la levure.
Contrairement aux gènes cellulaires, les gènes des virus herpes sont pour la plupart dépourvus d’introns Cela pourrait être un handicap à l’expression des gènes viraux dans la cellule hôte (rappelons au passage qu’il existe des gènes cellulaires dépourvus d’intron, donc non soumis à l’épissage, qui sont pourtant bien exprimés et échappent donc aux RNA-exosomes). Les virus herpes expriment un facteur dont le rôle est de favoriser l’accumulation cytoplasmique des ARNm viraux produits à partir de gènes dépourvus d’intron. L’étude du mode d’action d’une de ces protéines virales, EB2, a permis de mettre en évidence un nouveau rôle pour la protéine cellulaire SRSF3 (SRp20) impliquée dans l’épissage, l’export et la traduction de certains ARNm.
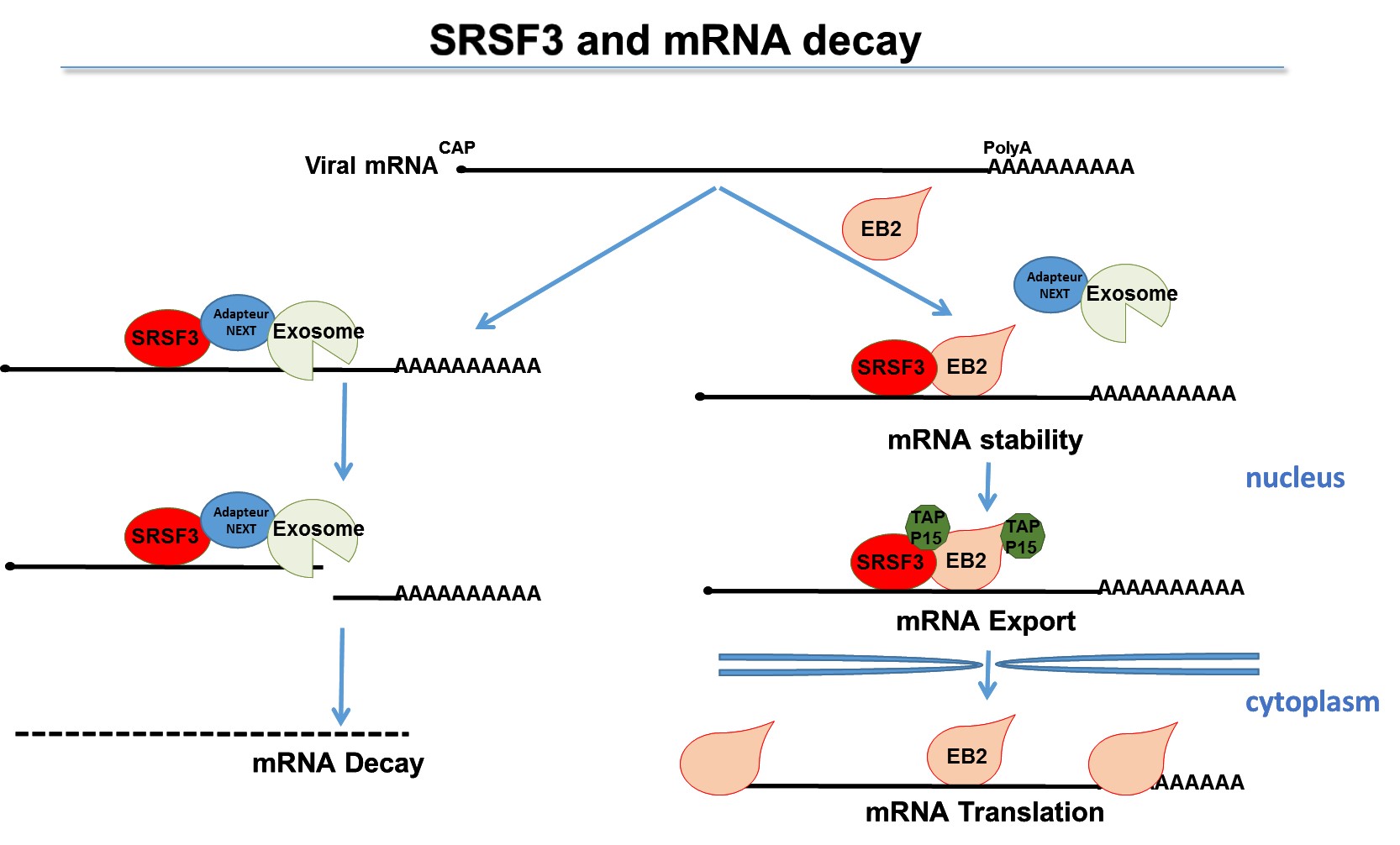
En absence de EB2, les ARN viraux produits à partir de gènes dépourvus d’intron ne sont pas stables et ils sont rapidement dégradés dans le noyau de la cellule par la voie du RNA-exosome. EB2 inhibe la dégradation de ces ARNm dans le noyau et permet leur export et leur accumulation dans le cytoplasme. La déplétion du complexe nucléaire RNA-exosome dans les cellules permet de stabiliser les ARNm viraux dans le noyau cellulaire.
Pour comprendre comment cette protéine virale peut stabiliser certains ARNm, une recherche de ses partenaires cellulaires a été réalisée et a permis d’identifier la protéine SRSF3. Comme dans le cas de la déplétion du complexe RNA-exosome, l’inhibition de l’expression de la protéine SRSF3 dans la cellule permet la stabilisation des ARNm viraux produits à partir des gènes dépourvus d’intron. La co-déplétion du complexe RNA-exosome et de SRSF3 ne permettant pas de stabiliser plus les ARNm viraux, l’hypothèse du recrutement du complexe par la protéine SRSF3 a été émise.
Le complexe RNA-exosome étant recruté sur ces ARN cibles par l’un des trois complexes adaptateur connus (TRAMP, PAXT ou NEXT), une interaction entre SRSF3 et les composants des complexes TRAMP et NEXT a été recherchée par co-immunoprécipitation. Les chercheurs ont montré une interaction entre la protéine SRSF3 et la protéine RBM7 du complexe NEXT suggérant le recrutement par SRSF3 lié à l’ARN cible, du complexe RNA-exosome par l’intermédiaire de la protéine RBM7 du complexe NEXT.
Ainsi, un lien entre la protéine SRSF3 du spliceosome et le RNA-exosome a pu être établi. L’interaction, déjà établie, entre EB2 (permettant la stabilisation et l’export des ARNm viraux) et la protéine SRSF3 permet probablement d’inhiber le recrutement du RNA-exosome et favorise la stabilisation nucléaire des ARNm viraux ainsi que leur export vers le cytoplasme où ils seront efficacement traduits en protéine, car la protéine virale permet d’accroitre très efficacement la production de ces protéines en favorisant le recrutement du complexe d’initiation de la traduction.
© Henri Gruffat
En savoir plus :
The splicing factor SRSF3 is functionally connected to the nuclear RNA exosome for intronless mRNA decay.
Mure F, Corbin A, Benbahouche NEH, Bertrand E, Manet E, Gruffat H.
Sci Rep. 2018 Aug 27;8(1):12901. doi: 10.1038/s41598-018-31078-1