Une enzyme pas comme les autres fait taire les séquences répétées du génome
Chez les plantes et les animaux, les protéines histones associées à l’ADN portent différentes modifications chimiques qui répriment soit l’expression des gènes, soit les éléments génétiques mobiles. Des chercheurs viennent de caractériser les propriétés inédites d’une enzyme de l’eucaryote unicellulaire Paramecium qui catalyse deux modifications silencieuses différentes et contrôle le silence des éléments transposables. Ces travaux, publiés dans la revue Nature Communications, révèlent que ces deux modifications partagent un rôle ancestral commun qui réduit au silence les éléments transposables.
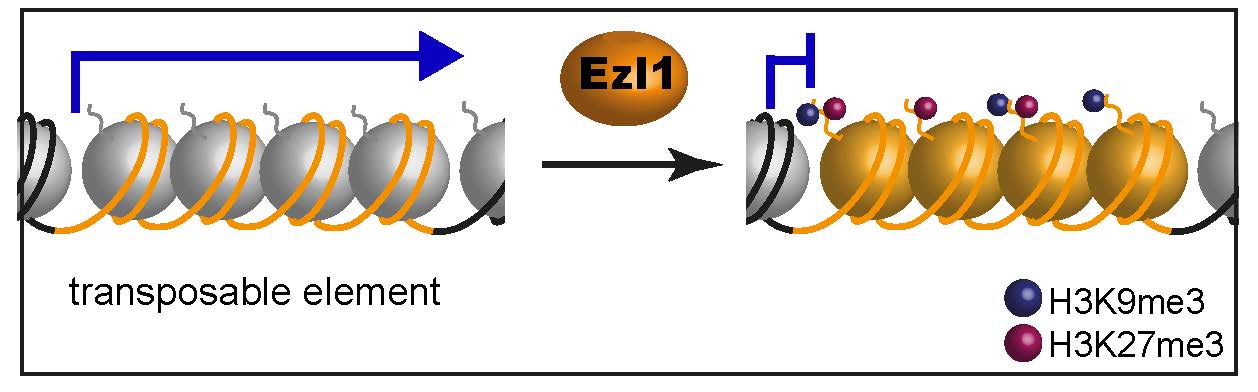
Le génome est compacté et organisé dans le noyau en complexe avec des protéines histones. Ce complexe ADN-protéines, appelé chromatine, est important pour le correct déroulement de nombreux processus cellulaires, en particulier le contrôle de l’expression des gènes. Les histones peuvent être l'objet de modifications chimiques, qui sont associées à des états héritables de l’expression des gènes. Parmi elles, la triméthylation de la lysine 27 de l’histone H3 ("H3K27") maintient réprimée l’expression de gènes durant le développement et joue un rôle crucial dans le contrôle épigénétique du développement. Une autre modification de l’histone H3, la triméthylation de la lysine 9 ("H3K9"), est quant à elle, associée à des régions génomiques silencieuses différentes, qui comportent, non pas des gènes, mais des séquences répétées, telles que des éléments transposables. Chez les plantes et les animaux, ces deux modifications silencieuses sont en général mises en place par des enzymes différentes.
Les chercheurs se sont focalisés sur la protéine Ezl1 de l’eucaryote unicellulaire Paramecium tetraurelia, homologue de la sous-unité catalytique du complexe Polycomb des mammifères. En combinant des approches biochimiques de tests enzymatiques in vitro et des expériences de génétique, ils ont montré que, de façon surprenante, l’enzyme Ezl1, bien que présentant des similarités de séquence et de structure tridimensionnelle avec la protéine humaine Ezh2, catalyse, en plus de son substrat canonique H3K27, la triméthylation de H3K9. Ces travaux montrent donc que l’activité de cette enzyme a une plus large spécificité de substrat que précédemment décrite pour ce type de protéines chez d'autres organismes. De plus, des expériences d’immunoprécipitation de la chromatine suivie de séquençage à haut débit montrent que ces deux modifications de l’histone H3 sont déposées dans les mêmes régions du génome par cette enzyme au cours du développement, et conduisent au silence des éléments répétés, une fonction généralement dévolue à la seule modification H3K9. Ces résultats suggèrent que ces deux modifications, souvent considérées comme ayant des fonctions distinctes, partagent une histoire évolutive commune et un rôle ancestral commun réduisant au silence les éléments transposables.
© Sandra Duharcourt
Pour en savoir plus :
The Polycomb protein Ezl1 mediates H3K9 and H3K27 methylation to repress transposable elements in Paramecium.
Frapporti A, Miró Pina C, Arnaiz O, Holoch D, Kawaguchi T, Humbert A, Eleftheriou E, Lombard B, Loew D, Sperling L, Guitot K, Margueron R, Duharcourt S.
Nat Commun. 2019 Jun 20;10(1):2710. doi: 10.1038/s41467-019-10648-5.