Un nouvel éclairage sur la ségrégation de l’ADN chez les bactéries
La machine moléculaire assurant la ségrégation de l’ADN chez les bactéries repose sur l’assemblage de plusieurs centaines de protéines ParB. Les chercheurs ont mis en évidence un mécanisme robuste d’assemblage stochastique de ce large complexe nucléoprotéique par l’isolement des complexes in vivo couplée au séquençage ADN à très haut débit et par la modélisation physico-mathématique de ces données. Ces travaux collaboratifs entre biologistes moléculaires et physiciens théoriciens expliquent comment l’ensemble des ParB se regroupe autour de seulement quelques sites spécifiques et ont été publiés dans la revue Molecular System Biology.
Le génome des bactéries est composé d’un ou plusieurs chromosomes et d’une quantité variable de petites molécules d’ADN autonomes appelées "plasmides". Ces derniers permettent aux bactéries de survivre et de se répandre dans des niches écologiques particulières, et sont très souvent porteurs des gènes de virulence ou de résistance aux antibiotiques. Après leur duplication, l’ensemble des différentes molécules d’ADN sont fidèlement transmises à chacune des deux cellules filles lors de l’étape de ségrégation.
Chez les bactéries, la ségrégation des chromosomes (à l'exception notable des gamma-protéobactéries), et des plasmides est assurée par un système actif minimaliste qui se compose de seulement trois éléments essentiels : un centromère (composé de courtes séquences d'ADN appelées parS), une protéine de fixation au centromère (ParB) et une protéine qui hydrolyse l’ATP (ParA), considérée comme la protéine motrice de la ségrégation. L’architecture du complexe nucléoprotéique, appelé complexe de partition, qui se forme sur le centromère, reste mal comprise et le mécanisme moléculaire conduisant à cet assemblage était controversé avec trois modèles en concurrence.
Les chercheurs du Laboratoire de Microbiologie et Génétique Moléculaires (LMGM-CBI, Toulouse), en collaboration avec l’équipe de physiciens théoriciens du Laboratoire Charles Coulomb (LCC, Montpellier) ont étudié le mécanisme de formation du complexe de partition du plasmide F d’Escherichia coli en combinant des approches de génétique moléculaire, de biochimie, de séquençage à haut-débit à l’échelle du génome entier, de microscopie à super-résolution et de modélisation physique.
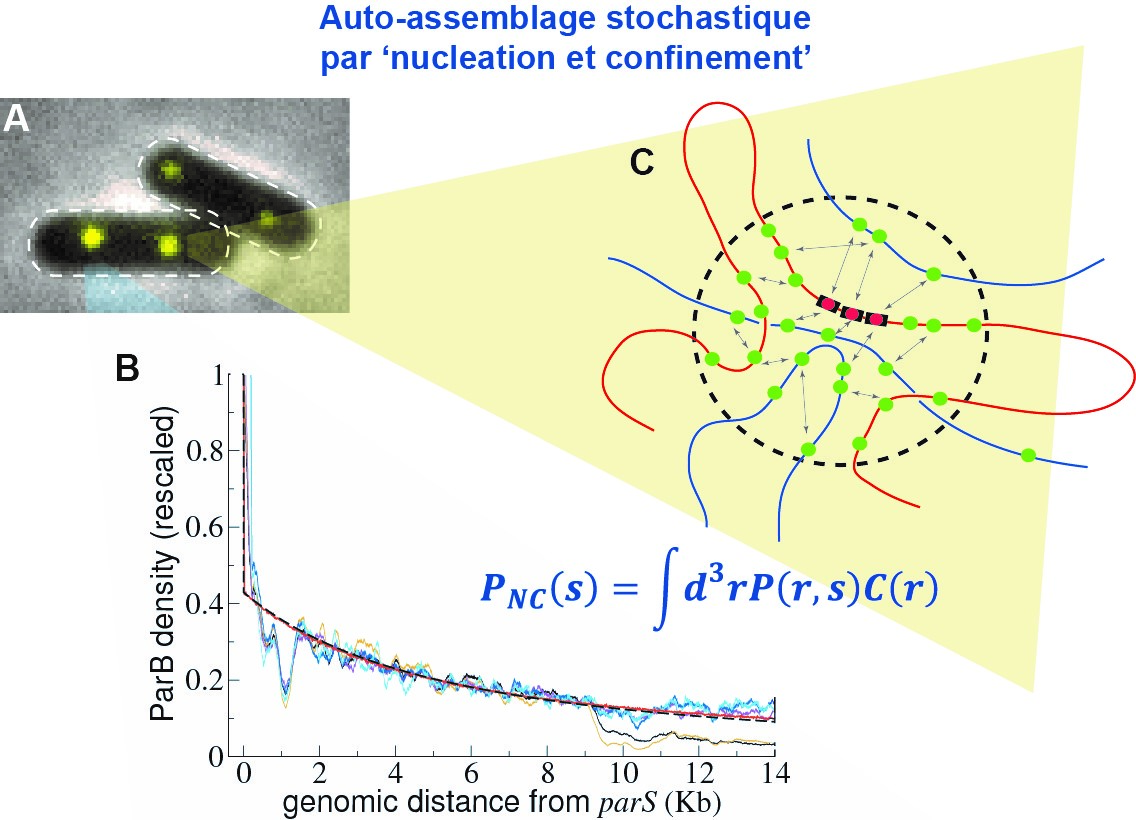
Afin de discriminer entre les trois mécanismes d’assemblage proposés, tous modélisés d’un point de vue physique, les chercheurs ont fait varier in vivo les paramètres clés du système de partition du plasmide F d’E. coli puis ont mesuré les effets de ces variations sur l'assemblage ParB/parS par ChIP-sequencing à très haute résolution (technique qui permet de connaitre précisément les sites de fixation de ParB). Les variations observées ont été confrontées aux prédictions théoriques de chacun des modèles révélant qu’un seul des modèles, appelé ‘Nucléation et confinement’, est compatible avec les données. De plus, conformément à la prédiction du modèle, ils ont montré par microscopie à haute résolution (FRAP) que ce complexe est très dynamique. Il résulte de multiples interactions synergiques, ParB-ParB et ParB-ADN non spécifique, qui confinent plus de 90% des ParB autour du centromère agissant comme point de nucléation. Après duplication de l’ADN, ce complexe dynamique sera séparé par la protéine ATPase partenaire, ParA, en deux complexes distincts qui seront positionnés de part et d’autre du septum de division.
Les chercheurs ont ensuite affiné la modélisation mathématique de ce modèle d’assemblage, puis étendu leurs résultats avec succès au système de partition de l’un des chromosomes de la bactérie pathogène Vibrio cholerae. L’ensemble des travaux montre que le mécanisme d’assemblage des complexes de partition est très robuste et a été conservé au cours de l’évolution. La ségrégation de l’ADN bactérien repose donc sur un auto-assemblage stochastique et dynamique impliquant un nombre restreint de petites séquences nucléotidiques.
© Jean-Yves Bouet
En savoir plus
A conserved mechanism drives partition complex assembly on bacterial chromosomes and plasmids.
Debaugny, R.E., Sanchez, A., Rech, J., Labourdette, D., Dorignac, J., Geniet, F., Palmeri, J., Parmeggiani, A., Boudscoq, F., Anton Leberre, V., Walter, J.-C. and Bouet, J.-Y.
Molecular Systems Biology (2018) 14, e8516 Published online 16.11.2018 DOI 10.15252/msb.20188516