Un mutant super-actif de l’ARN polymérase dédiée à la synthèse des ribosomes
L’ARN polymérase I (Pol I) synthétise les composants ARN des ribosomes qui assurent la production des protéines. Via diverses approches cellulaires, moléculaires, génétiques et biochimiques, les chercheurs ont isolé une polymérase mutante plus active que l’enzyme normale. L’existence de cette « super-enzyme » produisant plus d’ARN amène à de nouvelles façons d’envisager la régulation de la biogénèse des ribosomes. Ces résultats sont publiés dans la revue Plos genetics.
Les ribosomes, composés à la fois de protéines et d’ARN (ARNr), sont les machines moléculaires qui décodent les ARN messagers (ARNm) afin de synthétiser les protéines des cellules. Chez tous les eucaryotes, l'ARN polymérase I (Pol I) est une enzyme essentielle dédié à la synthèse des précurseurs de ces ARNr. A elle seule, la Pol I est responsable de plus de 60% de l’activité de synthèse totale des ARN d’une cellule en croissance. Ainsi, le niveau de production des ARNr est l’un des déterminants principaux du taux de croissance cellulaire, et en cela un excellent marqueur des cellules cancéreuses. En effet, de nouvelles molécules anticancéreuses montrent aujourd’hui que la Pol I est une cible privilégiée dans la lutte contre la prolifération des cellules malignes. Dans ce travail, les chercheurs ont utilisé l’organisme modèle des cellules eucaryotes, la levure de boulanger, Saccharomyces cerevisiae, qui est un outil puissant de par sa facilité de manipulation génétique, son génome entièrement séquencé, et sa croissance rapide.
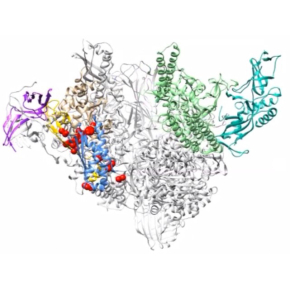
Les chercheurs ont utilisé un mutant de Pol I, dans lequel une sous-unité entière de l’enzyme (la sous-unité 49) est absente. Ce mutant est viable, mais possède de très forts défauts de synthèse d’ARNr et donc de croissance. Dans un premier temps, par une approche génétique, ils ont montré qu’il était possible de corriger ces défauts en ne modifiant qu’un seul acide aminé dans la Pol I délestée de la sous-unité 49. Plus d’une vingtaine de mutations ponctuelles différentes ont pu être ainsi identifiées (résidus en rouge sur la structure). De tels « double-mutants » possèdent une activité de synthèse d’ARNr et une croissance normale, alors même qu’une sous-unité entière de la Pol I est absente. Dans un second temps, les chercheurs ont étudié en détail la localisation des mutations sur la structure de la Pol I qui se retrouvent localisées dans une région bien précise de l’enzyme, à l’interface entre les domaines protéiques dits « mâchoire » et « Lobe » des deux plus grandes sous-unités de la Pol I. Ils proposent que la mobilité de ces domaines régule la fermeture du sillon dans lequel l'ADN s’insère afin que l’activité de l’enzyme s’effectue correctement.
Les chercheurs se sont enfin focalisés sur l’une de ces mutations (RPA135-F301S), et l’ont étudiée lorsque la Pol I n’est pas mutée/délestée de la sous-unité. Les Pol I mutées seulement pour cet acide aminé possèdent une activité de synthèse d’ARN supérieure aux ARN Pol I sauvages, tant in vitro qu’in vivo. Ces données renforcent l’idée que bien que la Pol I soit responsable de la majeure partie de la synthèse des ARN d’une cellule eucaryote, son activité doit être régulée négativement dans des conditions normales.
© Christophe Normand
Pour en savoir plus :
Genetic analyses led to the discovery of a super-active mutant of the RNA polymerase I.
Darrière T, Pilsl M, Sarthou MK, Chauvier A, Genty T, Audibert S, Dez C, Léger-Silvestre I, Normand C, Henras AK, Kwapisz M, Calvo O, Fernández-Tornero C, Tschochner H, Gadal O.
PLoS Genet. 2019 May 28;15(5):e1008157. doi: 10.1371/journal.pgen.1008157. eCollection 2019 May
Site du Laboratoire de biologie moléculaire eucaryote (LBM) : www-lbme.biotoul.fr