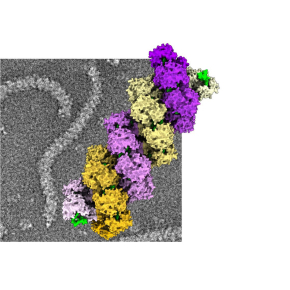
Un modèle du génome du virus de la grippe à l’échelle quasi-atomique
Dans un article publié dans la revue Science Advances, les scientifiques proposent un outil permettant d’observer les interactions entre le génome viral du virus de la grippe et les protéines qui lui sont associées avec une résolution de 5 Å soit 10-10 mètre (cinq ångström sont à un être humain ce qu’un être humain est au diamètre de 41 soleils). Ils ouvrent ainsi la voie vers une meilleure compréhension des mécanismes qui permettent l’assemblage et la réplication de ce virus sous haute surveillance.
Le virus de la grippe provoque chaque année des épidémies importantes touchant 2 à 6 millions de personnes en France[i]. Les souches des virus responsables de ces épidémies annuelles sont très proches des virus qui contaminent d’autres espèces, faisant craindre en permanence l’émergence de nouvelles formes par franchissement de la barrière inter-espèces. En effet, depuis octobre 2021, l'Europe subit l’épizootie d’influenza aviaire hautement pathogène (IAHP) la plus dévastatrice qu’elle ait jamais connue. Il s’agit d’une maladie virale hautement contagieuse qui affecte les oiseaux domestiques et sauvages. Les différentes souches grippales doivent donc rester en permanence sous très haute surveillance du fait du classement du virus influenza comme agent pathogène à haut potentiel pandémique par l’OMS.
Pour les scientifiques, il est tout aussi important de mieux comprendre les mécanismes pouvant les amener à muter ou à s’adapter à d’autres organismes. Dans cet article publié dans la revue Sciences Advances, les scientifiques ont mis au point une méthode permettant d’imaginer l’organisation de son génome à l’échelle atomique.
Ce génome est composé de huit molécules d’ARN simple brin. Chacune est recouverte de multiples copies de nucléoprotéines virales et leurs extrémités interagissent avec une ARN-polymérase (une sorte de photocopieuse essentielle à la prolifération virale) pour former le complexe ribonucléoprotéique (RNP). Extraites directement du virus et observées par microscopie électronique, les RNP ont une architecture complexe, extrêmement flexible et dynamique. Jusqu’à présent, leur observation par cryo-microscopie électronique (cryo-ME) n’avaient pas permis d’étudier le détail du positionnement de la nucléoprotéine avec l’ARN viral.
227 000 segments triés pour obtenir un modèle à 5 Å de résolution
Dans cet article publié dans la revue Science Advances, les scientifiques ont pris le parti de ne pas partir des complexes ribonucléoprotéiques purifiés à partir de virus. En s’appuyant sur leur expertise acquise lors des dix dernières années dans l’expression et la purification de la nucléoprotéine virale recombinante (c’est-à-dire produite in vitro), ils ont développé un protocole permettant d’auto-assembler des particules de type-RNP in vitro à partir de ces nucléoprotéines avec de petites sondes d’ARN. En développant cette approche, ils ont pu produire nettement plus de matériel biologique qu’en partant du virus, condition primordiale pour mener une étude à haute résolution par cryo-ME.
Chaque particule se retrouvant dans différentes orientations spatiales, il est donc nécessaire de disposer de grand jeu de données. Lors du traitement des données, les 227 000 segments de plus haute qualité ont été utilisés pour générer un premier modèle tridimensionnel à résolution nanométrique. Ce modèle souffrant de la flexibilité inhérente aux particules de type-RNP produites, un affinement localisé a été effectué permettant d’aboutir in fine à un modèle à 5 Å de résolution.
Une reconstruction 3D à haute résolution pour comprendre les mécanismes d’assemblage du virus et de sa réplication
Les scientifiques disposent maintenant d’une reconstruction 3D à haute résolution de ces particules similaires aux RNP grippales. Il devient ainsi possible de comprendre comment les molécules de nucléoprotéine interagissent entre elles au sein de cette architecture complexe flexible. Ce mode d’interaction est fortement similaire à celui observé dans les structures tridimensionnelles obtenues par diffraction des rayons X. Cette reconstruction tridimensionnelle à résolution subnanométrique obtenue par cryo-ME permet pour la première fois d’envisager l’agencement de l’ARN au sein des complexes ribonucléoprotéiques. Si l’ARN semble participer à la structuration de cette architecture complexe, il peut coulisser librement à la surface des protéines, sans doute pour pouvoir être facilement accessible pour l’ARN polymérase lors du cycle viral du virus de la grippe. Cette étude ouvre donc la possibilité de comprendre à terme les mécanismes qui permettent l’assemblage du virus et sa réplication.
[i] https://www.pasteur.fr/fr/centre-medical/fiches-maladies/grippe
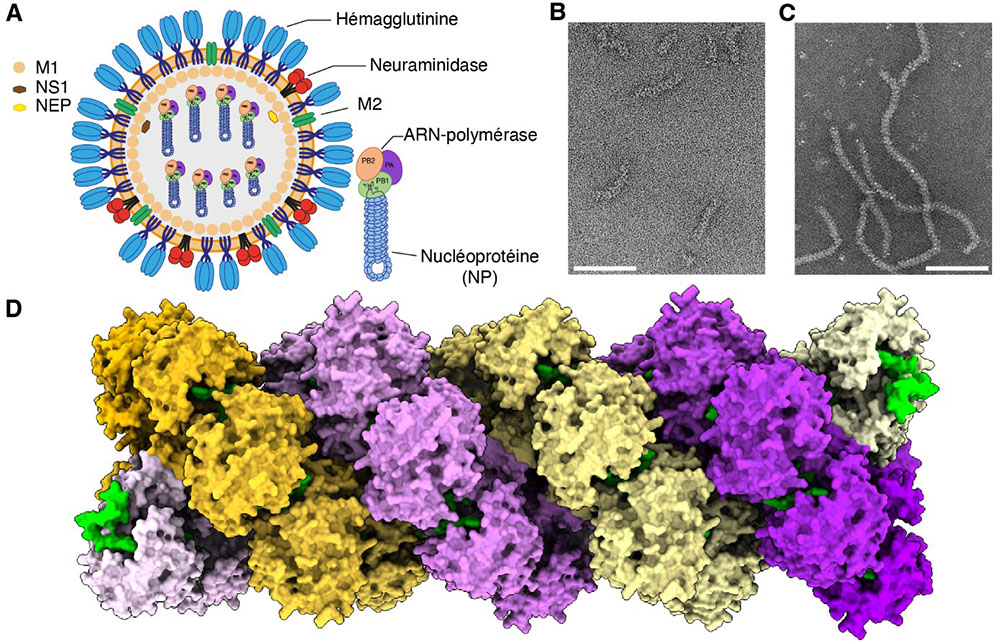
Figure : (A) Représentation schématique d'un virion du virus influenza et d’une RNP. Micrographes de (B) RNPs virales extraites de virus ou de (C) particules similaires aux RNPs assemblées in vitro à l'aide de NP recombinante et de petits ARN synthétiques. (D) Modèle antiparallèle d’encapsidation du génome grippal[DB1] [c2] généré à partir de la reconstruction à haute résolution des particules similaires aux RNPs. Les molécules de NP représentées en surface sont agencées en deux brins antiparallèles (colorées respectivement en un dégradé de jaune et de violet) et l'ARN est en vert, participe à la stabilisation de l’architecture. Les dégradés de couleur indiquent la polarité de chaque brin composant la RNP.
Florian Chenavier et al. Cryo-EM structure of influenza helical nucleocapsid reveals NP-NP and NP-RNA interactions as a model for the genome encapsidation.Sci. Adv.9,eadj9974(2023).DOI:10.1126/sciadv.adj9974
Contact
Laboratoire
Institut de biologie structurale - IBS (CNRS/CEA/Université Grenoble Alpes)
71 avenue des Martyrs
38042 Grenoble