Un mécanisme conservé de contrôle de la synthèse protéique dévoilé chez les plantes
TOR joue un rôle central dans la régulation de la synthèse des protéines chez les mammifères. Cette protéine est un facteur de croissance important également présente chez les plantes mais son mécanisme d’action dans la régulation traductionnelle globale n’était pas connue à ce jour. Dans une étude publiée dans Cell Reports, les scientifiques décrivent la découverte de protéines chez Arabidopsis agissant comme des répresseurs de la traduction sous le contrôle strict de TOR. Cette étude élucide comment TOR régule la traduction globale.
Mechanistic target of rapamycin (mTOR) est un hub cellulaire central intégrant les signaux intra- et extracellulaires d'énergie, de nutriments et d'hormones pour maintenir la synthèse des protéines, connue pour être le processus métabolique le plus énergivore. Cette synthèse des protéines se fait par la traduction des ARNm et est soumise à une régulation intense exercée principalement au niveau de l'initiation.
La principale voie d'initiation dépendante de la coiffe m7G en 5' des ARNm et du facteur d'initiation de la traduction 4E (eIF4E) liant la coiffe qui, en s’assemblant avec les protéines eIF4G et eIF4A, forme le complexe de liaison à la coiffe, nécessaire pour le chargement de la sous-unité ribosomique 40S.
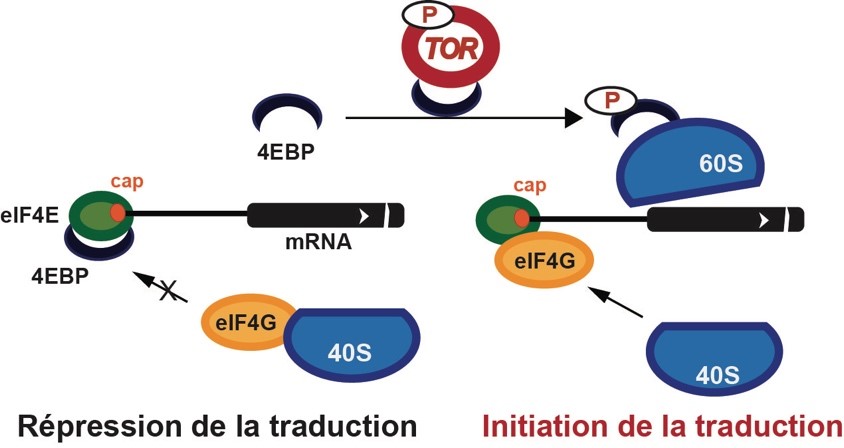
Chez les mammifères, l'initiation de la traduction dépendante de la coiffe est sous le contrôle négatif des protéines de liaison à l'eIF4E (4E-BP), qui interagissent avec l'eIF4E et bloquent le recrutement de la sous-unité ribosomique 40S sur l'ARNm. TOR inactive les 4E-BP en les phosphorylant, ce qui permet de stimuler de manière globale la traduction pour favoriser la croissance, la prolifération et la survie cellulaire. Ainsi, la dérégulation de la voie mTOR est souvent associée à une synthèse protéique incontrôlée, conduisant à diverses maladies chez l’Homme.
Chez les plantes, le rôle de TOR dans l'initiation de la traduction dépendante de la coiffe reste flou en raison de l'absence d'orthologues de 4E-BP. Inspirés par les larges possibilités de TOR, les scientifiques ont mené des recherches en utilisant des méthodes de génétique et de biologie moléculaire, et ont découvert chez la plante modèle Arabidopsis thaliana un ensemble de protéines capables d'exercer des fonctions suppressives similaires à celles des 4E-BP chez l’Homme. Ces 4E-BP-like (4EBP1/4EBP2) non conventionnelles participent au contrôle global de la traduction chez les plantes.
De plus, les scientifiques mettent en évidence plusieurs propriétés non canoniques et encore inconnues des 4EBP d'Arabidopsis. Le motif de liaison à l'eIF4E chez les mammifères commence par la tyrosine et est situé au milieu des 4E-BP, tandis qu'il apparaît à l'extrémité N-terminale des 4EBP chez les plantes, débutant par la phénylalanine, la tyrosine ou la leucine. Les auteurs se sont également demandés comment TOR phosphoryle les 4EBP alors qu’elles sont incapables d'interagir avec RAPTOR, le coactivateur de TOR, nécessaire à la phosphorylation chez les mammifères. Ils ont ainsi découvert que TOR peut interagir directement avec les 4EBP pour les phosphoryler.
De plus, il a été démontré que les 4EBP phosphorylées sont chargées sur les sous-unités 60S des ribosomes, qui servent de plateforme pour leur rétention dans les polysomes. Une fois que TOR est inactivé, les 4EBP sont déphosphorylées, dissociées des 60S, et transloquées vers la coiffe des ARNm via leur liaison à eIF4E pour bloquer l'initiation de la traduction. Ainsi, TOR régule l'activité des 4EBP par le biais d'événements de phosphorylation et d'échanges dynamiques entre les complexes ribosomiques et non ribosomiques dans le cytoplasme.
Chez Arabidopsis, TOR peut contrôler la division cellulaire en optimisant la traduction des ARNm clés des cyclines exprimées lors des phases G1 et G2 du cycle cellulaire. Les scientifiques ont utilisé différentes méthodes in planta dans des lignées d'Arabidopsis surexprimant 4EBP1 ou 4EBP2 pour déterminer leur capacité inhibitrice. Il a ainsi été démontré que 4EBP2 peut agir comme répresseur spécifique de la traduction des ARNm codant les cyclines D et B, suggérant que TOR inactive 4EBP2 pour favoriser la division cellulaire, l'activité méristématique et le taux de croissance global. De son côté, 4EBP1 peut agir au niveau de l'ajustement fin des réseaux métaboliques. En conclusion, ce travail démontre un nouveau rôle de TOR dans la régulation de l'initiation de la traduction dépendante de la coiffe chez les plantes grâce à des 4EBP atypiques, et met en évidence la diversité des processus de contrôle de la synthèse protéique entre les mammifères et les plantes.
En savoir plus :
Y. Dong, O. Srour, N. Lukhovitskaya, J. Makarian, O. Galzitskaya, D. Elser, M. Schepetilnikov and L.A. Ryabova (2023) Functional analogs of the mammalian 4E-BPs reveal a role for TOR in global plant translation, Cell Reports https://doi.org/10.1016/j.celrep.2023.112892
Contact
Laboratoire
Institut de biologie moléculaire des plantes (IBMP) UPR2357 du CNRS,
Université de Strasbourg, Strasbourg, France
