Un embryon virtuel pour cartographier l’activité des régions régulatrices du génome
Dans un article publié dans Nucleic Acids Research, des scientifiques décrivent spatial-scERA, une méthode combinant biologie moléculaire, séquençage en cellule unique et mathématiques, pour cartographier l’activité spatiale des régions régulatrices du génome, aussi appelées enhancers, dans un embryon virtuel 3D de drosophile. Cette approche ouvre de nouvelles perspectives pour explorer la régulation génétique in vivo.
Spatial-scERA : une approche interdisciplinaire pour localiser l’activité des enhancers
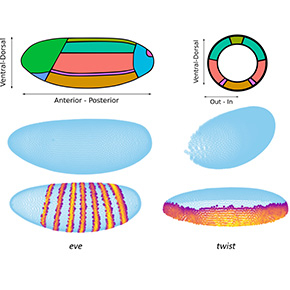
Le développement embryonnaire repose sur une régulation extrêmement précise de l’expression des gènes, à la fois dans le temps et dans l’espace. Parmi les éléments clés de ce processus figurent notamment les enhancers, de courtes séquences régulatrices d’ADN capables d’activer les bons gènes, au bon moment et au bon endroit. Mais cartographier leur activité, cellule par cellule, dans un embryon entier reste un défi de taille.
Pour y répondre, dans un article publié dans la revue Nucleic Acids Research, les scientifiques ont mis au point une approche originale, baptisée spatial-scERA, à l’interface de la biologie moléculaire, du séquençage du transcriptome en cellule unique et des mathématiques appliquées. Le principe ? Introduire dans des embryons de mouche du vinaigre (Drosophila melanogaster) des vecteurs contenant des enhancers associés à un gène « rapporteur », exprimé uniquement lorsque l’enhancer est actif. Les embryons sont ensuite dissociés en cellules individuelles, dont le transcriptome est analysé par séquençage en cellule unique (scRNA-seq). On obtient ainsi, pour chaque cellule, une double information : quels gènes y sont exprimés, et quel enhancer y est actif.
Visualiser la régulation génétique avec une résolution cellulaire
Mais comment replacer chaque cellule dans l’embryon d’origine une fois celui-ci dissocié ? C’est ici qu’intervient la dimension interdisciplinaire du projet. Grâce à une collaboration étroite entre biologistes, bioinformaticiens et mathématiciens, les scientifiques ont appliqué un algorithme issu de la théorie du transport optimal. Cette méthode a permis de reconstruire un embryon virtuel en 3D, dans lequel chaque cellule retrouve sa position approximative. Il est ainsi possible de visualiser précisément les régions où chaque enhancer est actif.
En combinant ces approches, l’équipe a pu dresser une carte spatiale de l’activité de 25 enhancers dans des embryons de drosophile, avec une résolution cellulaire. Même les enhancers actifs dans un nombre très restreint de cellules ont pu être localisés avec précision. Ces prédictions ont ensuite été validées par des expériences d’hybridation in situ. En comparant l’activité des enhancers avec celle des gènes exprimés dans l’embryon, à l’aide d’un score de similarité, ils ont également pu identifier les cibles génétiques probables de ces enhancers.
Avec spatial-scERA, il devient désormais possible d’explorer simultanément, dans un organisme multicellulaire, l’activité d’un grand nombre d’enhancers à l’échelle de la cellule unique. Robuste et adaptable, cette méthode ouvre de nouvelles perspectives pour l’étude de la régulation génétique pendant le développement. Elle pourrait être transposée à d’autres espèces animales ou à des systèmes comme les organoïdes, et bénéficier des progrès rapides en transcriptomique spatiale.
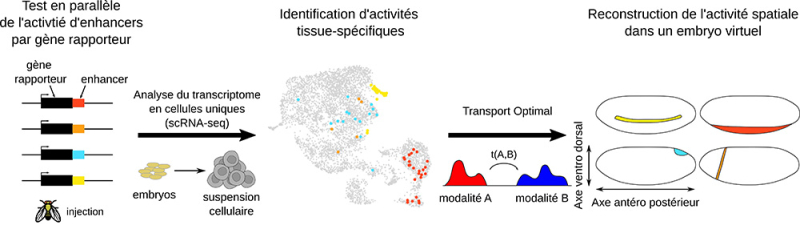
Figure : Illustration des étapes de la méthode spatial-scERA. Les différents enhancers à tester sont clonés individuellement dans des vecteurs contenant un gène rapporteur, exprimé uniquement lorsque l’enhancer est actif. Ces vecteurs sont injectés dans des embryons de drosophile, qui sont ensuite dissociés en cellules individuelles. Le séquençage du transcriptome permet de déterminer, pour chaque cellule, quel enhancer est actif et dans quel type cellulaire. Grâce à un algorithme de transport optimal, les cellules sont repositionnées dans un embryon virtuel 3D, révélant la distribution spatiale de l’activité des enhancers.
En savoir plus : Baptiste Alberti, Séverine Vincent, Isabelle Stévant, Damien Lajoignie, Hélène Tarayre, Juliette Mendes, Sergio Sarnataro, Paul Villoutreix, Yad Ghavi-Helm, Spatial-scERA: a method for reconstructing spatial single-cell enhancer activity in multicellular organisms, Nucleic Acids Research, Volume 53, Issue 14, 12 August 2025, gkaf684, https://doi.org/10.1093/nar/gkaf684
Contact
Laboratoire
Institut de génomique fonctionnelle de Lyon - IGFL (CNRS/ENS de Lyon)
32-34 avenue Tony Garnier
69007 Lyon