Un dialogue inédit entre protéines et ARN au cœur de la sporulation bactérienne
La sporulation permet à certaines bactéries de survivre dans des environnements hostiles. Dans un article publié dans mSystems, des scientifiques étudient, par une approche à haut débit, l’ensemble des protéines se liant à l’ARN : le RBPome. Ce dernier est profondément remodelé pendant la sporulation, ce qui suggère un rôle important de la régulation post-transcriptionnelle.
La sporulation : une stratégie de survie bactérienne
La sporulation est une stratégie de survie adoptée par certaines bactéries quand elles se trouvent dans un environnement hostile. Elles se différencient en spores dormantes capables de résister à des conditions extrêmes, aux techniques classiques de stérilisation et aux traitements antibactériens. Comprendre comment ce processus est régulé est essentiel pour répondre à des enjeux de santé publique, de sécurité alimentaire et pour développer de nouvelles applications industrielles.
Chez Bacillus subtilis, la bactérie modèle des bactéries sporulantes, la régulation de la sporulation est déjà bien décrite à deux niveaux : transcriptionnel, c’est-à-dire au niveau de l’ADN et traductionnel et post-traductionnel, au niveau des protéines.
En revanche, les mécanismes de régulation post-transcriptionnelle (au niveau des ARN) restaient largement inexplorés.
Une nouvelle approche pour explorer la régulation post-transcriptionnelle
Dans ce travail, publié dans la revue mSystems, des scientifiques ont adapté une méthode innovante, appelée OOPS (Orthogonal Organic Phase Separation) à une bactérie Gram positive.
Cette technique permet de capturer toutes les protéines qui interagissent avec des ARN (appelées protéines liant l’ARN pour RNA binding proteins ou RBP) et de suivre comment cet ensemble, appelé RBPome, évolue au cours de la sporulation. Cette méthode combine un « cross-link » UV in vivo qui stabilise les interactions ARN-protéine et une extraction acide guanidine-phénol-chloroforme pour isoler les complexes protéine-ARN.
Grâce à cette approche à haut débit, les scientifiques ont montré que le RBPome est profondément remodelé pendant la sporulation. Ce changement ne concerne pas seulement la quantité de protéines mais aussi leur capacité à se lier aux ARN.
Ces analyses ont permis d’identifier plus de 1 500 protéines liant l’ARN, dont certaines sont déjà connues, mais aussi de nombreuses nouvelles candidates.
Fait surprenant : plusieurs protéines métaboliques se sont révélées avoir une fonction inattendue d’interaction avec les ARN, illustrant la grande plasticité des réseaux de régulation chez les bactéries. Des protéines encore peu étudiées, comme KhpA et SpoVR, ont ainsi été identifiées comme se liant spécifiquement aux ARN durant la sporulation. Elles pourraient jouer un rôle clé dans l’adaptation environnementale
Ces résultats ouvrent une nouvelle voie pour comprendre comment les bactéries orchestrent leur différenciation et leur survie dans des environnements hostiles. Ils constituent une ressource précieuse pour de futures études mécanistiques et placent la régulation post-transcriptionnelle comme un niveau de contrôle essentiel et jusqu’ici peu connu de la sporulation.
En dévoilant ce nouveau niveau de régulation chez B. subtilis, ce travail propose un cadre inédit pour explorer la sporulation et l’adaptation bactérienne, avec des implications directes pour la microbiologie fondamentale et la lutte contre les pathogènes sporulants.
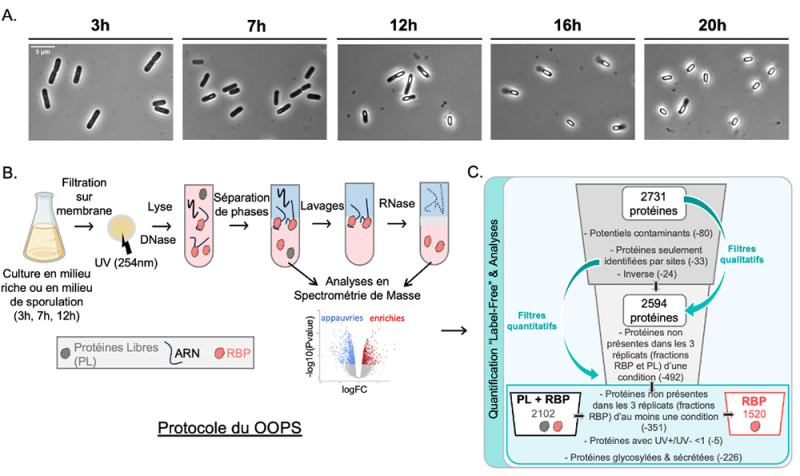
Figure : Caractérisation du RBPome de Bacillus subtilis pendant la sporulation
A : Suivi en microscopie de Bacillus subtilis pendant le processus de sporulation. B : Présentation du protocole du OOPS. Pour purifier le RBPome impliqué dans la sporulation, des expériences de OOPS ont été réalisées à 3, 7 et 12 h en milieu de sporulation (DSM). Les protéines libres (FP) et les RBP sont analysées (LC-MS) et quantifiées (LFQ). C : Filtres utilisés pour l'analyse des données protéomiques. Après utilisation de filtres quantitatifs et qualitatifs, sur les 2731 protéines répertoriées initialement, il reste 1520 RBP.
En savoir plus : Kaboré T, Belghazi M, Verthuy C, Galinier A, Delan-Forino C. Remodeling of RNA-binding proteome and RNA-mediated regulation as a new layer of control of sporulation. mSystems. 2025 Sep 23;10(9):e0049625. doi: 10.1128/msystems.00496-25. Epub 2025 Aug 15. PMID: 40815471; PMCID: PMC12456014.
Contact
Laboratoire
Laboratoire de chimie bactérienne - LCB (CNRS/Aix-Marseille Université)
31 Chemin Joseph Aiguier
13009 Marseille - France
