Thérapies antitumorales et médecine régénérative : identification de nouveaux régulateurs de la plasticité cellulaire
Les changements de l’identité et de la plasticité cellulaire jouent un rôle majeur au cours de la reprogrammation cellulaire vers l’état pluripotent. En parallèle, ces modifications émergent également comme des régulateurs importants de différentes étapes de la transformation oncogénique. Une meilleure compréhension des mécanismes moléculaires associés constitue donc un enjeu crucial, à la fois pour la médecine régénérative mais aussi pour la biologie du cancer. Pourtant, les séquences d’évènements qui coordonnent ces changements d’identité et de plasticité n’ont jamais été disséquées de manière comparative dans des cellules en cours de reprogrammation et de transformation. Une nouvelle étude publiée dans la revue Nature Cell Biology contribue à lever le voile sur ces interrogations.
Dans cette étude, les scientifiques ont comparé, à l’échelle de la cellule unique, les trajectoires de cellules qui commencent à se reprogrammer pour devenir des cellules souches pluripotentes induites (iPS) et celles de cellules qui commencent à se transformer pour devenir cancéreuses. On sait depuis plusieurs années que les processus de reprogrammation et de transformation oncogénique présentent un certain degré d’analogie puisqu’ils sont tous deux stochastiques et limités par des barrières telles que la mort cellulaire et la senescence. Cependant, l’approche comparative menée par les chercheurs, combinée à des analyses bio-informatiques des modifications du transcriptome et de l’épigénome, a permis de révéler l’existence d’un programme moléculaire intrinsèque commun à ces deux « destins » cellulaires.
De plus, en s’attardant sur les étapes précoces des deux processus, les scientifiques ont démontré que la plasticité et l’identité cellulaire sont contrôlées par des réseaux moléculaires au moins partiellement indépendants. En effet, l’utilisation d’une nouvelle combinaison de marqueurs (Bcl11b et Thy1) a permis la capture d’états cellulaires intermédiaires qui émergent très précocement au cours des deux processus. La caractérisation fonctionnelle et multi-omique de ces états a permis de montrer qu’ils avaient déjà acquis un certain degré de plasticité cellulaire alors que leur identité restait inchangée. Au niveau moléculaire, cette approche a ainsi conduit à l’identification d’un axe de régulation, centré sur le facteur de transcription Atoh8, qui bloque l’acquisition de plasticité cellulaire et donc la reprogrammation et la transformation. Ici aussi, les auteurs ont montré que cet axe c-Myc/Atoh8/Sfrp1 contrôle spécifiquement la plasticité des cellules sans impacter leur identité.
Cette étude permet donc de mieux appréhender le contrôle de l’identité et de la plasticité cellulaire dans un contexte de régénération ou de développement cancéreux. Conceptuellement, elle ouvre également la voie à différentes approches. Les chercheurs de cette étude s’intéressent désormais au design de stratégies de régénération qui seraient non tumorigéniques dans le cadre d’applications en médecine régénérative. De plus, ces travaux indiquent la possibilité de développer des stratégies anti-tumorales qui cibleraient spécifiquement et exclusivement la plasticité cellulaire.
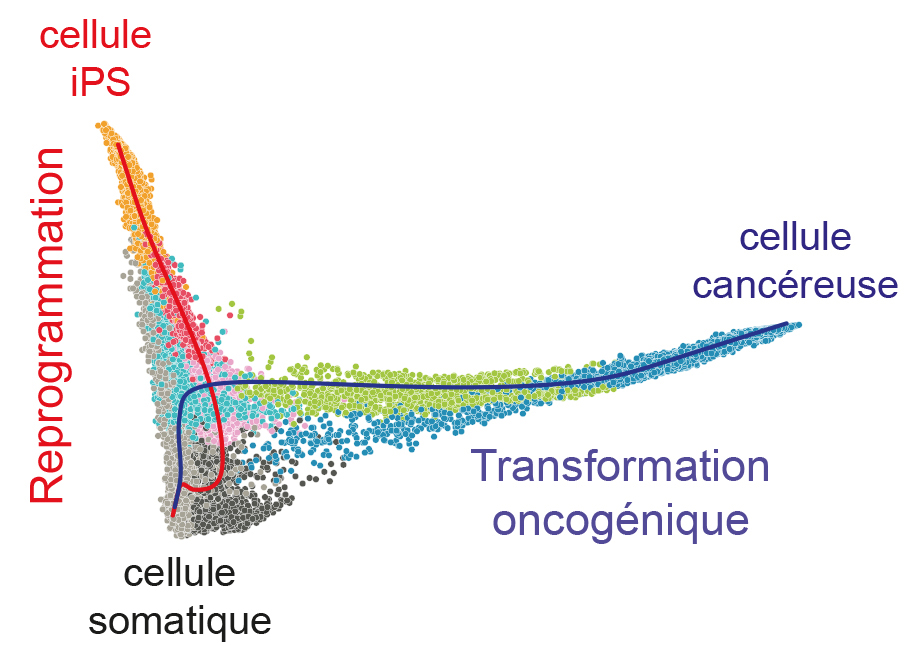
Figure : Trajectoire de cellules en cours de reprogrammation ou de transformation oncogénique. Le graphe présente des données transcriptomiques à l’échelle de la cellule unique de cellules somatiques (fibroblastes embryonnaires) induites à reprogrammer en cellules iPS (par l’expression exogène des facteurs de transcription Oct4, Sox2, Klf4 et c-Myc) ou à devenir cancéreuses par l’expression de c-Myc et d’une forme oncogénique de K-Ras (K-RasG12D). Les trajectoires reconstruites sont présentées sous forme de ligne rouge pour la reprogrammation et bleue pour la transformation. Elles intersectent avant de bifurquer, indiquant un programme commun. Chaque point correspond au transcriptome d’une cellule unique. La couleur des points correspond à des clusters de cellules présentant des similitudes transcriptomiques.
Pour en savoir plus :
Comparative roadmaps of reprogramming and oncogenic transformation identify Bcl11b and Atoh8 as broad regulators of cellular plasticity
Huyghe, G. Furlan, J. Schroeder, E. Cascales, A. Trajkova, M. Ruel, F. Stüder, M. Larcombe, Y. Bo Yang Sun, F. Mugnier, L. De Matteo, A. Baygin, J. Wang, Y. Yu, N. Rama, B. Gibert, J. Kielbassa, L. Tonon, P. Wajda, N. Gadot, M. Brevet, M. Siouda, P. Mulligan, R. Dante, P. Liu, H. Gronemeyer, M. Mendoza-Parra, J. M. Polo & F. Lavial
Nature Cell Biology, 8 septembre 2022. doi:10.1038/s41556-022-00986-w
Contact
Laboratoire
Centre de recherche en cancérologie de Lyon (Inserm/CNRS/Université Lyon1)
Centre Léon Bérard
28 rue Laennec
69008 Lyon