Structure atomique des complexes de réplication virale associés aux membranes
Les structures de la protéine 1 non structurale (nsP1) du virus du Chikungunya, obtenues par cryo-microscopie électronique, révèlent les mécanismes qui sous-tendent l'association de la machinerie de réplication virale avec les organites membranaires induits par le virus au sein des cellules hôtes. Cette structure révèle des détails sans précédent sur les mécanismes de la liaison membranaire, de l'assemblage des protéines et de l'activation de l'activité catalytique d'un virus à ARN. Ces travaux sont publiés dans la revue Nature.
Le virus Chikungunya, et d'autres virus à ARN tels que le Dengue ou le Coronavirus, installent leurs machineries de réplication sur les membranes internes des cellules infectées, ce qui conduit à la production des génomes viraux. La protéine non structurelle 1 (nsP1) du Chikungunya est responsable de cette invasion des membranes et est également capable de marquer les ARN viraux pour leur camouflage parfait, similaires aux ARNm cellulaires. De cette façon, le virus passe inaperçu dans les premières étapes de l'infection et trompe les cellules pour qu'elles produisent les protéines virales. Une fois que la cellule se rend compte qu'elle est infectée, le virus a déjà envahi toute la cellule et il est trop tard pour l'arrêter.
Dans le cadre d'une collaboration avec le service de microscopie cryo-électronique du centre national espagnol de biotechnologie, les chercheurs ont analysé la structure de cette protéine nsP1 du virus du Chikungunya. Sa structure révèle que l'attachement aux membranes se fait par la formation de macro-assemblages constitués de douze protéines identiques, en forme de pore. Ces pores ouvrent l'entrée des compartiments viraux où les virus "dissimulent" leurs génomes à la réponse immunitaire cellulaire. Les pores contrôlent l'accès aux organelles de réplication virale et assurent le marquage de l'ARN à sa sortie vers le cytoplasme cellulaire. L'analyse fine de la structure révèle des détails sans précédent sur les mécanismes de la liaison membranaire, de l'assemblage des protéines et de l'activation de l'activité catalytique d'un virus à ARN. Ainsi, les chercheurs ont constaté que la protéine virale nsP1 est inactive sous sa forme monomèrique et que ce n'est que lorsqu'elle est assemblée sur les membranes cellulaires que l'activité est déclenchée ce qui permet à la protéine de "faire le bon travail au bon endroit".
L'élucidation de cette structure redéfinit les complexes de réplication du virus Chikungunya comme de véritables réacteurs de réplication, en principe capables de traiter simultanément douze molécules d'ARN, ce qui pourrait expliquer la forte infectivité de ces virus. Les détails atomiques étonnants de cette structure pourraient ouvrir la voie à la conception de stratégies ciblant les protéines virales dans le contexte de leur association membranaire donc, finalement, dans leur état naturel actif.
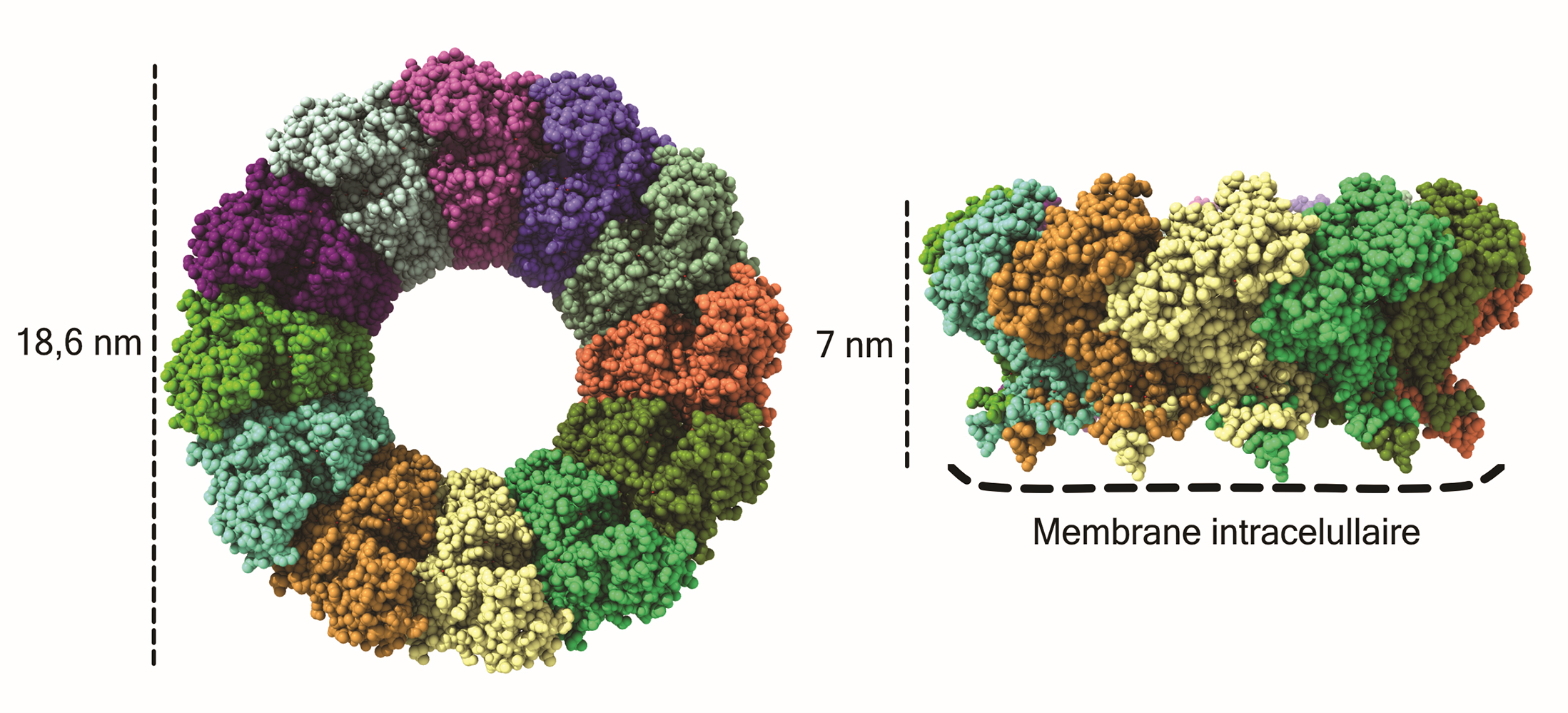
Figure : Structure atomique des pores nsP1 du virus du Chikungunya avec les protéines nsP1 représentées dans différentes couleurs. Chaque atome est représenté par une sphère. Sur la gauche, le pore est représenté en vue frontale et à droite vue latérale. Les dimensions du pore sont indiquées à gauche et les sites de contact avec la membrane sont indiqués en bas.
Pour en savoir plus :
Capping pores of alphavirus nsP1 gate membranous viral replication factories
Jones R, Bragagnolo G, Arranz R and Reguera J
Nature 16 Dec 2020 DOI: 10.1038/s41586-020-3036-8.
Contact
Laboratoire
Architecture et Fonction des Macromolécules Biologiques (AFMB) - (Aix-Marseille Univ/CNRS)
Campus Luminy - Case 932
163 Avenue de Luminy
13288 Marseille Cedex 09 France