Résurrection d’états ancestraux d’une enzyme permettant la vie à haute salinité
Les Haloarchées sont des microorganismes qui se développent dans un environnement saturé en sels imposant des contraintes majeures à leurs protéines. La "résurrection" de protéines ancestrales est une approche de plus en plus utilisée pour explorer l’adaptation des protéines aux changements de leur environnement. Les travaux publiés dans la revue Molecular Biology and Evolution, détaillent comment 3 lignées d’haloarchées ont indépendamment adapté la solubilité et la stabilité d’une de leurs enzymes à des conditions de salinité extrêmes.
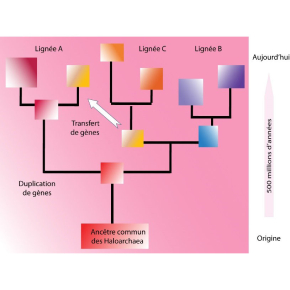
Les archées (Archaea) forment l’un des trois groupes fondamentaux d’organismes qui peuplent la terre, les deux autres étant les bactéries et les eucaryotes, ce 3° groupe réunissant les organismes dont les cellules sont pourvues d’un noyau. Les haloarchées sont un groupe d’archées adaptées aux environnements hypersalins tels que les lacs salés. Ce sont ces espèces qui donnent, à certaines périodes de belles couleurs roses aux marais salants. Diverses études suggèrent que l’origine des Haloarchées remonte à 500 millions d’années. Il a été récemment établi que les haloarchées suivent la stratégie « salt-in », c’est-à-dire que l’intérieur de leur unique cellule est aussi concentré en sels que l’est le milieu extérieur, la stratégie alternative étant de pomper hors des cellules les ions salins. Cette stratégie « salt-in » nécessite des adaptations spécifiques à la plupart des protéines de ces organismes puisque des concentrations salines élevées détruisent la structure tridimensionnelle et donc la fonctionnalité des protéines.
Il est intéressant de comprendre la nature de ces adaptations moléculaires, et comment elles se sont mises en place au cours de l’histoire évolutive des haloarchées. Une équipe pluridisciplinaire de biochimistes, biologistes de l’évolution et bio-informaticiens s’est donc réunie pour aborder cette question via la "résurrection" de protéines ancestrales (estimation bioinformatique de leurs séquences, synthèse chimique de leurs gènes, biosynthèse puis purification et caractérisation fonctionnelle). Cette technique, en place depuis quelques années, est spécialement appropriée à l’analyse de l’histoire évolutive d’une ou plusieurs protéines.
Dans un premier temps, des méthodes statistiques établissent une estimation bio-informatique des états ancestraux les plus probables d'un ensemble de séquences protéiques homologues (donc issues d'un ancêtre commun) présentes dans les haloarchées actuelles. Il était nécessaire de reconstituer l’histoire évolutive des protéines étudiées depuis la divergence de ce dernier ancêtre commun, ce qui s’est révélé complexe en raison de plusieurs duplications et transferts horizontaux de gènes (le transfert horizontal de gènes est un phénomène au cours duquel un organisme absorbe de l’ADN provenant d’un autre organisme sans en être le descendant).
Des fragments d’ADN dont la séquence code ces protéines ancestrales sont alors synthétisés puis introduits dans des bactéries pour produire ces protéines ancestrales. Celles-ci sont purifiées, leur activité enzymatique ainsi que leur stabilité conformationelle sont mesurées. On peut ainsi faire correspondre l’effet des mutations accumulées sur une longue période évolutive dans une enzyme avec les changements de propriété qu’elle a subis. Comme la structure tridimensionnelle, déterminée par cristallographie aux rayons X, d’une de ses enzymes existe, il est possible de localiser précisément les zones où agissent les mutations.
Cette démarche a été appliquée à l’étude d’une enzyme impliquée dans le métabolisme, la malate déshydrogénase (MalDH). Ces analyses ont établi que l’enzyme du dernier ancêtre commun à toutes les MalDH d’haloarchées était déjà enrichie en acides aminés acides, et que cette propriété qui permet une grande solubilité a été conservée tout au long de l’histoire des haloarchées. Il a été également établi que la MalDH a acquis, de façon indépendante dans trois lignées différentes, la capacité à conserver sa structure tridimensionnelle grâce à des ions stabilisant son assemblage oligomérique, et donc son activité, sur une large gamme de concentrations salines et de pH.
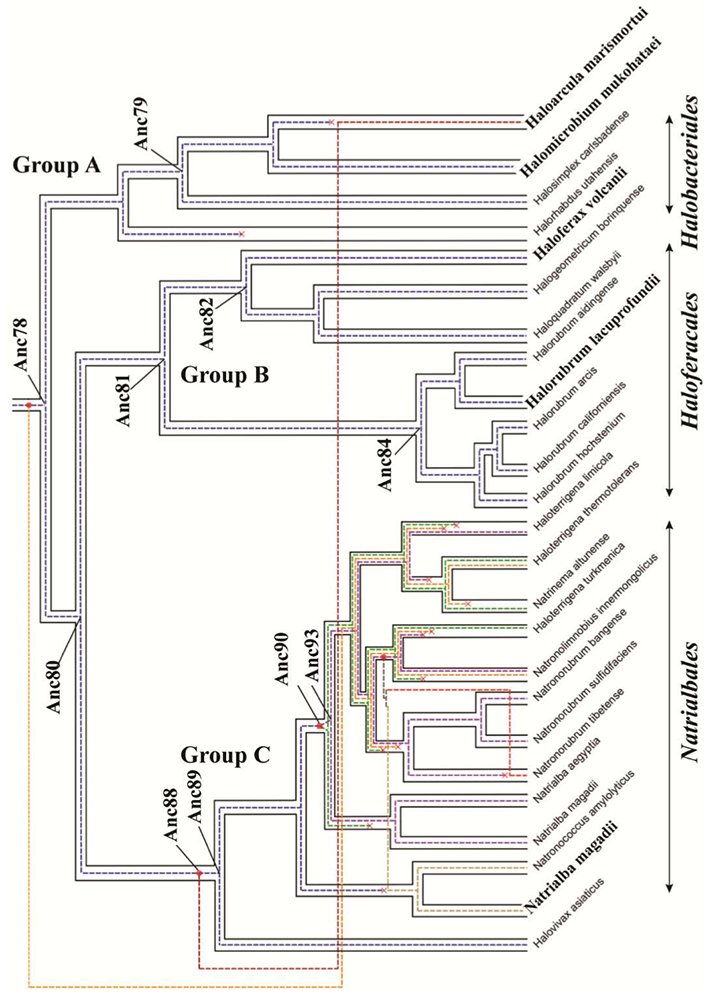
Figure : Ce schéma exprime simultanément les parentés évolutives des haloarchées étudiées (les tubes) et celles du ou des gènes MalDH présents dans ces organismes (lignes pointillées colorées à l’intérieur des tubes). Deux phénomènes font que ces deux types de parenté doivent être distingués : 1) une duplication génique (carré rouge marqué Anc90), 2) les transferts horizontaux de gènes qui apportent à un organisme un gène provenant d’une autre lignée évolutive (lignes pointillées extérieures aux tubes). Les croix indiquent des pertes de gènes. Les étiquettes Anc## indiquent les 10 organismes ancestraux dont les séquences ont été estimées, synthétisées et étudiées.
Pour en savoir plus :
Resurrection of Ancestral Malate Dehydrogenases Reveals the Evolutionary History of Halobacterial Proteins : Deciphering Gene Trajectories and Changes in Biochemical Properties
Samuel Blanquart, Mathieu Groussin, Aline Le Roy, Gergely J Szöllosi, Eric Girard, Bruno Franzetti, Manolo Gouy & Dominique Madern.
Molecular Biology and Evolution 11 Mai 2021. https://doi.org/10.1093/molbev/msab146
Contact
laboratoire
Institut de biologie structurale [CNRS/Université Grenoble Alpes/CEA]
71 avenue des Martyrs
CS 10090
38044 Grenoble Cedex 9