Réparer l’ADN cassé : comment les polymérases X sauvent nos cellules
L’ADN de nos cellules subit en permanence des lésions, dont la plus grave est la cassure simultanée des deux brins qui le composent. Dans une étude, publiée dans Nature Communications, des scientifiques révèlent en détail comment des enzymes qui copient l’ADN participent à la réparation de ce type de lésion. A terme, la connaissance intime des acteurs intervenant dans ce mécanisme pourra permettre de le manipuler afin de rendre les cellules cancéreuses plus sensibles aux thérapies dont le mode d’action est de casser l’ADN.
Les cassures double-brin de l’ADN : une menace permanente pour nos cellules.
L’ADN qui constitue notre patrimoine génétique est constamment endommagé par des agents chimiques ou physiques présents dans l’environnement. L’un des dommages le plus grave, la cassure double-brin, survient lorsque les deux brins de la molécule d’ADN sont rompus. Cette lésion peut être mortelle pour une cellule si elle ne parvient pas à la réparer.
Cependant les cellules humaines disposent d’une voie de réparation très efficace, la jonction d’extrémités non homologues (NHEJ). Ce mécanisme soude entre elles les extrémités de la cassure et peut même nettoyer des extrémités abimées pour faire un ajustage parfait avant leur soudure. Il était déjà connu que certaines enzymes qui copient l’ADN, les ADN polymérases, participaient à cette étape d’ajustement. Mais leur mode d’action précis restait mal compris.
Une interaction clé révélée entre Ku et les polymérases X
Dans les cellules humaines, la réparation par NHEJ débute par la liaison rapide du complexe protéique Ku en forme d’anneau sur les extrémités cassées. De façon remarquable l’anneau Ku sert de point d’ancrage commun aux différents complexes protéiques qui assurent toutes les étapes suivantes de la NHEJ.
Dans une étude publiée dans la revue Nature Communications, des scientifiques viennent d’élucider comment les ADN polymérases de la famille X participent à la réparation des cassures double-brin par NHEJ. Cette étude dévoile plusieurs structures à l’échelle atomique, obtenues par cryo-microscopie électronique montrant les interactions entre les ADN polymerases X et Ku sur une cassure de l’ADN. Ces structures mettent en évidence un site d’interaction spécifique entre Ku et le domaine BRCT, un motif commun à toutes les polymerases X.
Pour prouver l’importance de cette interaction, les scientifiques ont introduit des mutations ciblées sur la zone de contact entre Ku et le motif BRCT. Ils ont ensuite évalué les conséquences de ces changements sur la capacité des cellules à réparer les cassures de l’ADN. Pour cela, ils ont construit un outil original qui permet de mesurer l’activité de copie sur les extrémités d’une molécule d’ADN cassée en un point précis et introduite dans les cellules. Les résultats confirment que le domaine BRCT des ADN-polymérases X est indispensable au recrutement sur les cassures et à l’activité des polymérases X, et donc à la survie des cellules en cas de coupure double-brin.
Une avancée vers des thérapies anticancéreuses plus ciblées.
Les traitements comme la radiothérapie et d’autres thérapies anticancéreuses visent justement à induire dans les cellules tumorales de nombreuses cassures double-brin, pour provoquer un stress létal. Une meilleure connaissance du fonctionnement du mécanisme NHEJ et de tous ses acteurs ouvre la voie à des stratégies thérapeutiques plus précises, permettant par exemple de fragiliser sélectivement les cellules cancéreuses en perturbant leur capacité à réparer l’ADN.
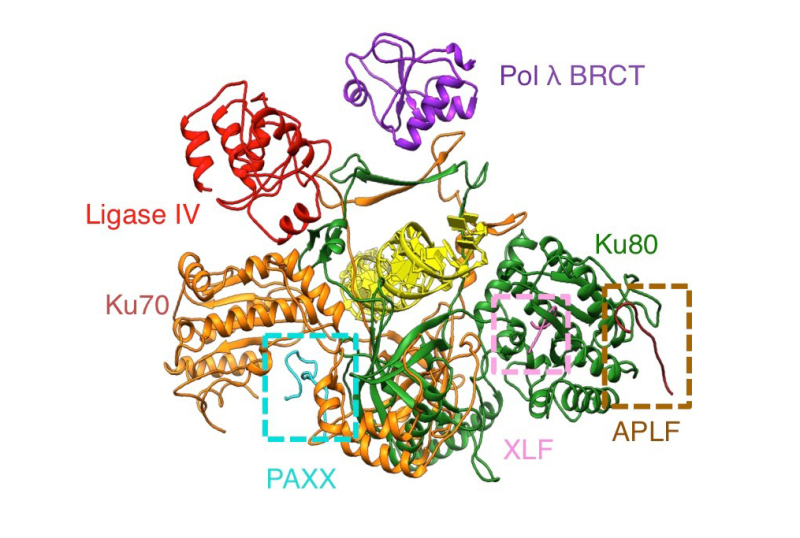
Figure : L’anneau Ku70/80 (orange/vert) autour de l’ADN (jaune) constitue un point d’ancrage pour la liaison de Pol λ (domaine BRCT, violet), Ligase IV (domaine BRCT1, rouge), PAXX (domaine P-KBM, cyan), XLF (domaine X-KBM, rose) et APLF (domaine A-KBM, brun), protéines intervenant dans la réparation des cassures double-brin de l’ADN par NHEJ.
En savoir plus : Frit, P., Amin, H., Zahid, S. et al. Structural and functional insights into the interaction between Ku70/80 and Pol X family polymerases in NHEJ. Nat Commun 16, 4208 (2025). https://doi.org/10.1038/s41467-025-59133-2
Contact
Laboratoire
Institut de pharmacologie et biologie structurale - IPBS (CNRS/Université de Toulouse)
205 route de Narbonne
31077 Toulouse

