Régler le réveil du génome embryonnaire
Dans un embryon en développement, il est essentiel de contrôler le timing de l’activation des gènes. Par ailleurs, à chaque division cellulaire, les programmes développementaux des cellules mères doivent être fidèlement transmis à leurs cellules filles. Afin d’étudier ces processus de timing d’expression et de mémoire, les chercheurs ont utilisé l’embryon précoce de drosophile où il est aisé de suivre en temps réel le réveil du génome zygotique. Dans une étude multidisciplinaire publiée dans la revue Nature Communications, ils ont pu montrer comment un facteur de transcription (Zelda) régulait quantitativement le tempo de l’activation du génome au cours du développement embryonnaire précoce.
Le développement harmonieux d’un organisme pluricellulaire nécessite un contrôle spatio-temporel de l’expression des gènes, ainsi qu’une transmission fidèle de cette expression au cours de multiples divisions.
En utilisant l’embryon de drosophile comme organisme modèle, il est possible de suivre en temps réel le réveil du génome dans un embryon vivant et l’héritabilité de cette information génétique à chaque division cellulaire. Ainsi, une mémoire de l’activité transcriptionnelle entre un noyau "mère" et ses "filles" (issues de la division du noyau mère) a récemment pu être visualisée et quantifiée dans l’embryon de drosophile. En quantifiant les temps d’activation à la 13eme division (nc14) du zygote, un biais clair a pu être identifié entre les noyaux dont les mères étaient transcriptionnellement actives au cycle précédent et les noyaux voisins issus de mères inactives. Ce biais dans les temps d’activation est une mesure de la mémoire mitotique.
Afin de comprendre quels sont les mécanismes régulant cette mémoire, les chercheurs se sont intéressés à l’activateur transcriptionnel Zelda, un facteur pionnier aux fonctions clés lors des premières heures de développement de la drosophile. En quantifiant précisément les temps d’activation de plusieurs transgènes possédant un nombre croissant de sites de fixation de Zelda, les chercheurs ont démontré que Zelda modulait le timing de la transcription (Figure A). En distinguant les temps d’activation de noyaux provenant de mères actives de ceux issus de mères inactives, les chercheurs ont montré que Zelda accélérait le tempo de ces deux types de populations et tendait de ce fait à supprimer le biais de mémoire mitotique.
Un modèle mathématique a été développé et a permis de mieux appréhender le rôle de Zelda sur le nombre et la durée des étapes nécessaires à l’activation de la transcription. Les temps d’attente précédant une activation de la transcription sont ici modélisés par une série de transitions entre des états épigénétiques discrets, tel un escalier (Figure B). Les variables du modèle sont le nombre et la durée de chaque transition. Le modèle ainsi que les données biologiques suggèrent que Zelda agit principalement sur la durée (et non le nombre) des étapes de pré-initiation de la transcription.
En analysant la cinétique de la protéine Zelda au cours du cycle cellulaire via des expériences d’imagerie quantitatives (Fluorescence Recovery After Photobleaching (FRAP) et Fluorescence Correlation Spectroscopy (FCS)), les chercheurs ont pu révélerque Zelda n’était pas retenu pendant la mitose, mais que ce facteur pionnier était très dynamique, ne se liant à la chromatine que pendant quelques secondes in vivo. Cependant cette liaison transitoire pourrait être compensée par une augmentation locale de sa concentration. En effet les chercheurs ont démontré que Zelda s’accumulait au sein de microenvironnements nucléaires (‘hubs’). L’accumulation de Zelda dans ces microenvironnements favoriserait une coopérativité entre plusieurs facteurs de transcription et/ou le rapprochement de plusieurs séquences d’ADN cibles (enhancers). L’ensemble de ces travaux, issus d’une recherche interdisciplinaire et hautement collaborative (CNRS-IGMM, CNRS-IRIM, Université de Montpellier et Université du Wisconsin à Madison) ouvre des nouvelles pistes pour mieux comprendre le comportement dynamique de la machinerie responsable de l’activation des gènes et de l’organisation nucléaire durant le développement embryonnaire.
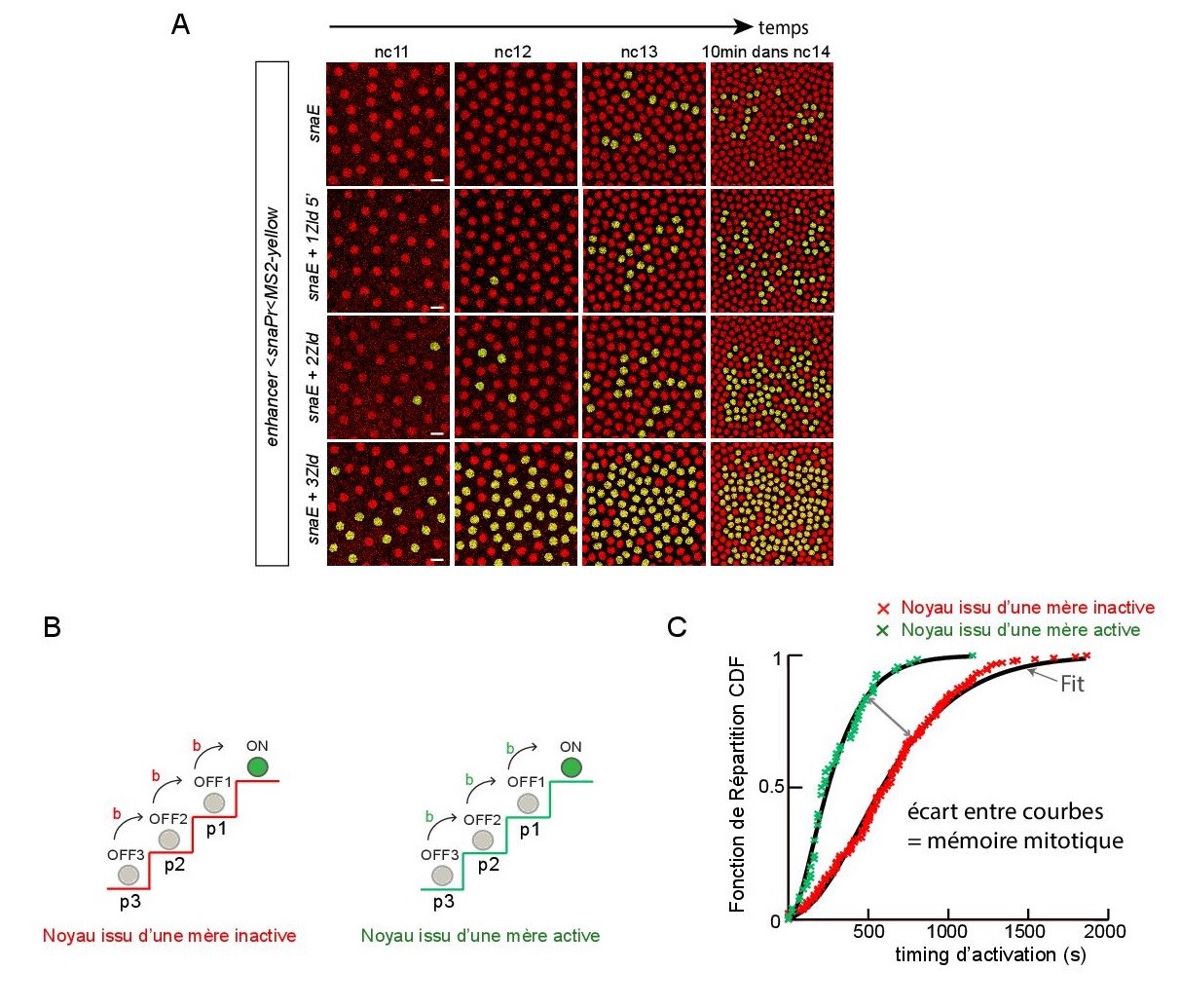
B) Représentation schématique des états discrets (OFF1, OFF2 et OFF3) nécessaires pour atteindre l’activation de la transcription (état ON), lorsqu'ils proviennent d'un noyau mère inactif (escalier rouge) ou d’un noyau mère actif (escalier vert). Après la mitose, la probabilité «p» qu'un noyau initie le processus d'activation de la transcription à partir d'un état donné est fonction des paramètres p1, p2 et p3 (p1 = p (OFF1), p2 = p (OFF2) et p3 = p (OFF3)). La durée de chaque transition d’un état à l’autre est fournie par le paramètre «b». Les paramètres de ce modèle sont prédits séparément pour les noyaux provenant de mères actives et inactives.
C) Fonction de Répartition (Fonction de distribution cumulative (cdf)) des temps d’activation au nc14. Les données des temps d’activation de noyaux résultants de mères actives sont représentées en vert et celles issues de mères inactives sont représentées en rouge. Les paramètres du modèle sont estimés à partir d’un ajustement aux données (courbes noires).
© Mounia Lagha
vidéo
Vidéo : Vidéo obtenue par imagerie confocale d’un embryon de drosophile vivant, filmé depuis le cycle nucléaire 10 jusqu’à la fin du cycle nucléaire 14 lorsque les futures cellules musculaires (progéniteurs du mésoderme) s’invaginent dans l’embryon via le processus de gastrulation. Les noyaux sont marqués en rouge (Histone-RFP) et les ARNm neo-synthétisés sont marqués en vert (MCP-GFP). Dans cet exemple, l’activation rapide de la transcription est détectée grâce au système MS2/MCP à partir d’un transgène contenant trois sites de fixations pour la protéine Zelda (snaE+3Zld<MS2). Cette vidéo a été accélérée, les images originales ont une résolution temporelle de 1 image toutes les 24 secondes.
© Mounia Lagha
Audiodescription
En savoir plus
-
Temporal control of gene expression by the pioneer factor Zelda through transient interactions in hubs.
Dufourt J, Trullo A, Hunter J, Fernandez C, Lazaro J, Dejean M, Morales L, Nait-Amer S, Schulz KN, Harrison MM, Favard C, Radulescu O, Lagha M.
Nat Commun. 2018 Dec 5;9(1):5194. doi: 10.1038/s41467-018-07613-z.
