Réarrangements génomiques en cascade induits par des intermédiaires toxiques de réparation de l’ADN
Les cassures de l’ADN rompent l’intégrité du chromosome et de l’information génétique qu’il contient. Dans un article publié dans la revue Genes and Development des scientifiques ont approfondi la caractérisation d’un mécanisme à risque pour la stabilité du génome. Ces résultats permettront de mieux comprendre des réarrangements chromosomiques révélés par les nouvelles techniques de séquençage dans divers organismes et dans certaines situations pathologiques.
Une cassure de l’ADN rompt l’intégrité du chromosome et de l’information génétique qu’il contient. La fidélité du processus de réparation des cassures par recombinaison homologue participe au maintien de la stabilité du génome. Les scientifiques ont récemment identifié un mécanisme pathologique issu de la recombinaison homologue, nommé Multi-invasion-Induced Rearrangement (MIR) capable à partir d’une cassure sur un chromosome d’induire une translocation entre deux chromosomes indépendants intacts.
Dans cette publication, les scientifiques ont développé une nouvelle méthodologie extrêmement sensible permettant de révéler la présence d’un réarrangement chromosomique rare dans une population de cellules. En couplant cette technologie avec d’autres méthodes de génétique moléculaire, ils ont révélé que ce processus de MIR possède en réalité deux voies qui affectent différemment la stabilité du génome. Une première voie, MIR2, est bénigne : elle génère des insertions de séquences entre deux chromosomes sans opérer de translocation. Elle requiert une synthèse d’ADN substantielle et n’est applicable que dans des contextes où les chromosomes intacts partagent des séquences en commun.
L’autre voie, MIR1, est plus délétère : elle génère une translocation et des cassures supplémentaires dont la réparation conduit à une cascade de réarrangements pouvant se propager à d’autres chromosomes. Cette voie est active dans n’importe quel contexte de séquence, et ne requiert pas de synthèse d’ADN à partir d’une matrice double-brin. Elle se produit après un arrêt prolongé du cycle cellulaire sur des intermédiaires de réparation persistants. Elle illustre comment la voie de la recombinaison homologue, souvent considérée comme à haute-fidélité, peut contribuer à la formation d’importants réarrangements du génome à partir d’une seule lésion initiale.
Grace à ces résultats, les scientifiques proposent une signature mutationnelle unique que ce mécanisme est susceptible de laisser dans le génome à proximité de séquences répétées. Cette signature guidera à l’avenir l’interprétation des origines de réarrangements médiés par les séquences répétées des génomes eucaryotes que la technologie émergente de séquençage à long brins commence à révéler.
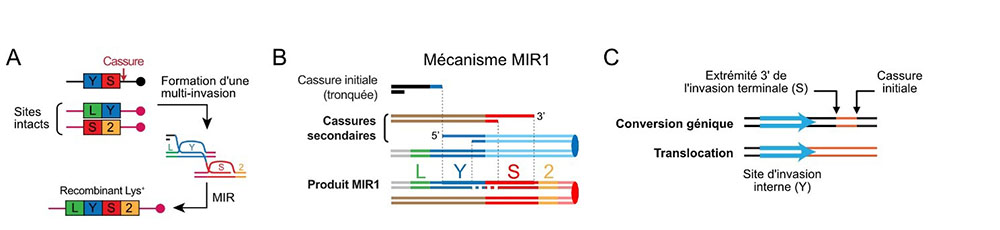
Figure : A) Système expérimental permettant de sélectionner un réarrangement chromosomique induit par le MIR à la suite de la formation d’une cassure induite à proximité de la séquence YS. B) Spécificité du mécanisme MIR1, qui conduit à une translocation et à la formation de deux cassures supplémentaires, susceptibles d’engendrer une cascade de réarrangements de génome.C) Exemple de signature de séquence du mécanisme MIR1, impliquant deux chromosomes.
En savoir plus :
Delineation of two multi-invasion-induced rearrangement pathways that differently affect genome stability
Reitz D, Djeghmoum Y, Watson RA, Rajput P, Argueso JL, Heyer WD*, Piazza A*.
Genes and Development, 4 août 2023, DOI: 10.1101/gad.350618.123
Contact
Laboratoire
Laboratoire de biologie et modélisation de la cellule - LBMC (CNRS / ENS Lyon)
ENS Lyon – Site Monod
46 Allée d’Italie
69007 Lyon