Pourquoi les ARN polymérases III ne déambulent-elles pas dans les génomes ?
Une terminaison de la transcription efficace est cruciale non seulement pour la synthèse correcte de tous les ARN cellulaires par les ARN polymérases mais aussi pour assurer que ces enzymes restent confinées à leurs sites de fonction. Dans un article publié dans la revue Science Advances, les scientifiques dévoilent un nouveau mécanisme de terminaison qui empêche les ARN polymerases de type III de déambuler dans le génome, ce qui pourrait mettre en danger d'autres processus tels que la réplication ou la réparation de l'ADN.
Chez les organismes eucaryotes, la transcription est effectuée par trois ARN polymérases (ARNpols) distinctes qui sont spécialisées dans la production de différentes classes de transcrits et qui semblent adopter des stratégies différentes pour terminer leur transcription. L'ARN polymérase III (ARNpol III) est responsable de la synthèse de transcrits très abondants, tels que les ARN de transfert et l'ARN ribosomal 5S, qui sont absolument nécessaires à la prolifération cellulaire. Le modèle traditionnel de la terminaison de la transcription par l’ARNpol III postule que cette dernière peut terminer de manière efficace et précise sur une séquence d'ADN spécifique sans avoir besoin de protéines accessoires. Cependant, ce modèle s’est avéré trop simple. Dans cette étude réalisée chez la levure Saccharomyces cerevisiæ, les scientifiques montrent que les mécanismes qui conduisent à la terminaison de la transcription par l’ARNpol III sont bien plus complexes.
En utilisant une approche génomique de pointe permettant de cartographier les ARNpols III en cours de transcription avec une très haute résolution, ils observent qu’une bonne fraction des ARNpols III ne respecte pas les signaux de « stop » à la fin des gènes et continue de transcrire les régions en aval. Ils montrent également que la terminaison de ces ARNpols III qui ont échappé au terminateur canonique est assurée par une voie « de secours » qui dépend d’une protéine évolutivement très conservée, l'ARN hélicase Sen1, à laquelle avait été attribuée précédemment un rôle majeur dans la terminaison de la transcription des deux autres ARNpols eukaryotes (ARNpol I et l’ARNpol II). En utilisant un système in vitro avec des composants hautement purifiés pour analyser la terminaison de manière contrôlée, ils montrent que Sen1 peut directement induire la terminaison de la transcription par l’ARNpol III.
Ces résultats révèlent donc l'existence d'un mécanisme universel de terminaison reposant sur un facteur protéique conservé qui opère au niveau des trois ARNpols eucaryotes malgré leurs propriétés et leur composition en sous-unités différentes. L’ensemble de ces données permet de mettre en évidence la manière dont les organismes ont développé des voies complémentaires pour empêcher les ARNpols d'envahir les régions génomiques au-delà des gènes qu’elles transcrivent.
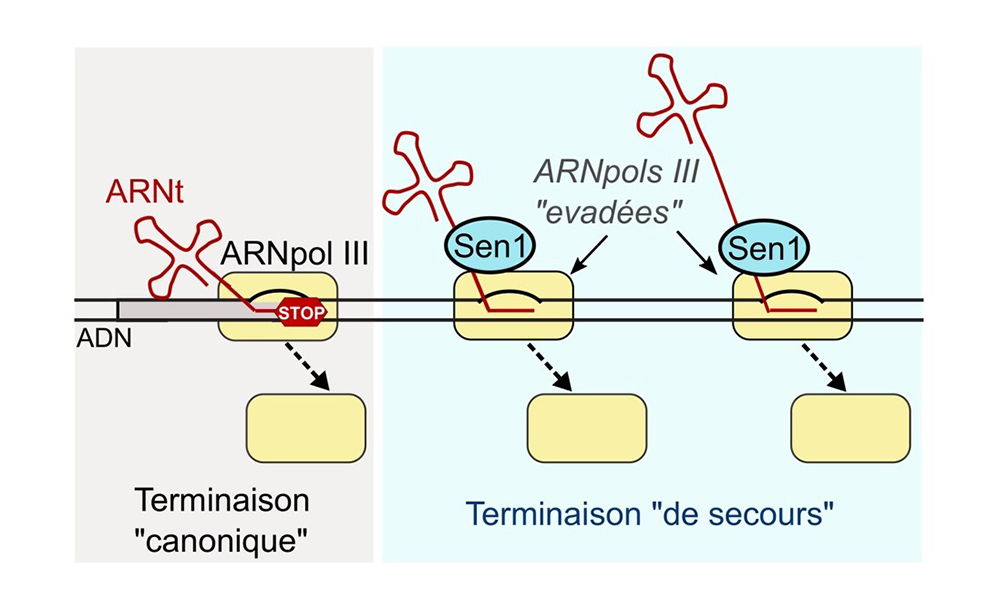
Figure : Modèle des voies alternatives de terminaison de la transcription par l’ARNpol III. La terminaison de la transcription au niveau du terminateur canonique à la fin des gènes n’est que partiellement efficace. Les ARNpols III qui ont échappé à cette première voie de terminaison sont ciblées pas l’ARN hélicase Sen1, qui induit leur relâchement de l’ADN, protégeant ainsi les régions génomiques environnantes.
Pour en savoir plus :
An integrated model for termination of RNA polymerase III transcription
Juanjuan Xie, Umberto Aiello, Yves Clement, Nouhou Haidara, Mathias Girbig, Jana Schmitzova, Vlad Pena, Christoph W. Müller, Domenico Libri and Odil Porrua.
Science Advances 13 juillet 2022. DOI: 10.1126/sciadv.abm9875
Contact
Laboratoire
Institut Jacques Monod (Université Paris Cité/CNRS)
15 rue Hélène Brion
75013 Paris