Moteur contrôlant la taille des boucles d’ADN sur les chromosomes
L’organisation tridimensionnelle (3D) des génomes présente de profonds remaniements lors du cycle cellulaire afin d’assurer le bon fonctionnement de processus biologiques tels que la transcription, la réparation de l’ADN ou encore la ségrégation des chromosomes. La cohésine, un complexe protéique annulaire, est l’un des acteurs clé à la base de cette organisation dans de nombreuses espèces. Les scientifiques ont caractérisé les mécanismes moléculaires qui contrôlent le positionnement des boucles de chromatine le long des chromosomes par la cohésine. Ces résultats sont publiés dans la revue Nature Structural and Molecular Biology.
La cohésine est un complexe protéique formant un anneau, de la famille des complexes de maintenance de la structure des chromosomes (SMCs). De la bactérie à l’homme, ces anneaux protéiques sont capables d’établir et de maintenir des boucles de chromatine le long des chromosomes. Sur la base de résultats obtenus in vitro sur des molécules d’ADN, il a été proposé que les complexes SMCs capturent de petites boucles d’ADN à travers leur anneau puis les élargissent progressivement grâce à leur activité ATPasique, ce qui entraînerait leur déplacement le long de la molécule d’ADN. Chez les mammifères, la cohésine forme et maintient de telles boucles pendant l’interphase du cycle cellulaire. De récentes études ont montré que des variations de la position et de la taille de ces boucles influencent et/ou régulent des processus tels que la transcription, la recombinaison ou encore la réparation de l’ADN. Toutefois, les moteurs moléculaires contrôlant l’agrandissement et le positionnement de ces boucles le long des chromosomes restent mal définis.
Pour identifier le moteur de l’extrusion de l’ADN, les scientifiques ont utilisé une approche de capture de conformation de chromosomes (Hi-C), permettant de caractériser le repliement des chromosomes dans des souches de levure Saccharomyces cerevisiae déplétées de manière contrôlée en différentes protéines du complexe cohésine, et soigneusement synchronisées à différents moments du cycle. En disséquant les effets de ces déplétions, ils ont révélé que le facteur Scc2/NIPBL2, dont on sait qu’il est nécessaire pour charger les cohésines sur l’ADN, est également requis pour promouvoir l’agrandissement des boucles d’ADN. Scc2/NIPBL2 joue ce rôle actif de moteur moléculaire en stimulant l’activité « ATPasique » nécessaire au déplacement des cohésines sur l’ADN. De plus, les scientifiques ont montré que la fonction activatrice de Scc2 est réprimée par la liaison de la protéine auxiliaire Pds5 aux cohésines. Cette inhibition du moteur moléculaire par Pds5 a comme conséquence de maintenir et stabiliser les boucles d’ADN à des positions spécifiques le long des chromosomes. Il restait alors à déterminer comment le complexe cohésine passe d’une forme dynamique à une forme stable pendant l’expansion des boucles. Les auteurs ont pu démontrer que l’acétylation de la sous-unité Smc3 par l’acétyl transférase Eco1 favorise la fixation de Pds5, au détriment de Scc2, inhibant ainsi l’expansion des boucles.
Ces résultats ont été conjointement publiés avec une étude du laboratoire du Dr Benjamin Rowland (Chromosome biology, NKI) montrant que ces mécanismes sont conservés dans les cellules humaines.
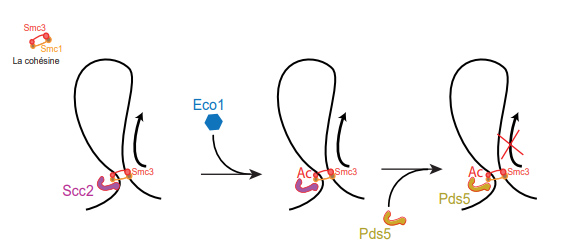
Figure : Régulation de l’expansion des boucles d’ADN du génome par les sous-unités du complexe cohésine. Scc2 stimule l’activité d’extrusion de l’ADN au travers de l’anneau de cohésine. L’acétylation de la sous-unité Smc3 par Eco1 conduit à la stabilisation de Pds5 sur la cohésine et par conséquent à l’inhibition de l’expansion de la boucle d’ADN.
Pour en savoir plus :
Smc3 acetylation, Pds5 and Scc2 control the translocase activity that establishes cohesin-dependent chromatin loops.
Bastié N, Chapard C, Dauban L, Gadal O, Beckouët F, Koszul R.
Nat Struct Mol Biol. 16 juin 2022. doi: 10.1038/s41594-022-00780-0.
The cohesin acetylation cycle controls chromatin loop length through a PDS5A brake mechanism.
van Ruiten MS, van Gent D, Sedeño Cacciatore Á, Fauster A, Willems L, Hekkelman ML, Hoekman L, Altelaar M, Haarhuis JHI, Brummelkamp TR, de Wit E, Rowland BD.
Nat Struct Mol Biol. 16 juin 2022 doi: 10.1038/s41594-022-00773-z.
Contact
Laboratoire
Equipe Organisation du génome
Centre de biologie intégrative (CBI), Unité MCD (Université de Toulouse III - Paul Sabatier/CNRS)
118 route de Narbonne 31000 Toulouse
Unité Régulation Spatiale des Génomes
Département Génomes et Génétique (CNRS/Institut Pasteur)
Institut Pasteur
28 rue du Dr. Roux
75724 Paris Cedex 15
