Modification des protéines : la myristoylation décodée à l’échelle de l’organisme
Une équipe de l'Institut de biologie intégrative de la cellule, en collaboration avec l’Institut de chimie des substances naturelles et l’Ecole Polytechnique, a cartographié et quantifié pour la première fois, chez l’homme et la plante modèle Arabidopsis thaliana, le myristoylome, c’est-à-dire l’ensemble des protéines portant une étiquette constituée d'un acide gras qui cible ces protéines aux membranes. Ces données publiées dans les revues Nature Chemical Biology et Plant Cell permettent enfin d’élucider comment la machinerie de modification décrypte ses cibles.
Toute protéine subit des modifications, réversibles ou irréversibles, qui impactent son cycle de vie et sa fonction. Le nombre de modifications décrit pour une seule protéine peut varier de 0 à 100 et chaque espèce modifiée constitue une protéoforme. Plus de 400 modifications différentes contribuent à cette diversité. On estime que les génomes humains et des plantes qui contiennent environ 20 000 gènes peuvent générer plusieurs milliards de protéoformes, les modifications assurant ainsi une diversité extrêmement importante.
Parmi les modifications protéiques, la myristoylation (MYR) correspond à un des dispositifs les plus intrigants que les cellules utilisent pour positionner précisément les protéines dans des compartiments membranaires spécifiques. La modification consiste à ajouter à une extrémité d'une protéine une petite étiquette faite d'un acide gras appelé myristate. Cette étiquette confère un caractère huileux qui agit à la fois comme « code postal » et comme ancre aidant à « expédier » la protéine vers une membrane particulière tout en assurant la fixation stable autant de temps que nécessaire. Bien que cette étiquette soit cruciale pour la survie cellulaire et fondamentale dans des processus importants pour la santé et les pathologies, la MYR est l'une des modifications les plus difficiles à étudier et à décoder, le répertoire complet de MYR - le myristoylome - étant inconnu jusqu'à présent.
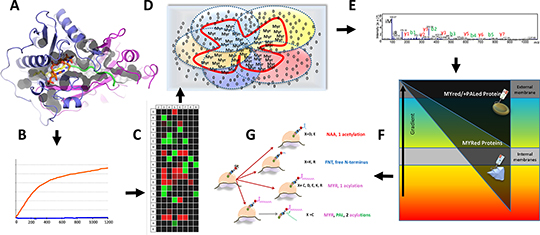
Une étude de l'équipe de Carmela Giglione à l'I2BC à Gif-sur-Yvette réalisée en collaboration avec deux autres équipes du plateau de Saclay (ICSN et Ecole Polytechnique) a réussi à cartographier pour la première fois des ensembles complets de protéines possédant une étiquette de MYR, et ce chez l’homme et le modèle de plante Arabidopsis thaliana. Ils ont surmonté un certain nombre d’obstacles critiques liés à l'analyse de la MYR en développant et en appliquant progressivement des approches complémentaires. Tout d’abord, les structures cristallines de l'enzyme humaine impliquée dans cette modification ardue associée à plusieurs cibles connues ont révélé des sites de liaison inattendus ; ceci a permis de comprendre le schéma général de reconnaissance du dispositif de la modification, resté insaisissable jusqu’alors. Les informations obtenues par ces données structurales ont été ensuite combinées à (i) la mise en place d’un test reconstitué permettant de mesurer à très grande échelle la MYR éventuelle d’une bibliothèque de mimes de toutes les protéines cibles possibles de la cellule, (ii) la mise au point d’algorithmes dédiés utilisant ces données et (iii) une analyse globale de la diversité des protéines subissant cette modification réalisée chez les deux organismes pour valider ces approches.
Les auteurs ont aussi réalisé en parallèle un profilage adapté des protéines membranaires pour révéler leur niveau de MYR. Ce protocole implique un fractionnement cellulaire combiné à une séparation à haute résolution des protéines avec l’identification des composants huileux par spectrométrie de masse couplée à des méthodes d'acquisition dépendant des données ciblant spécifiquement les peptides modifiés par MYR. Ce travail a révélé la distribution relative et quantitative de la plus grande partie des protéines avec MYR de la cellule. L’étude représente également le plus grand ensemble de données de protéines avec MYR identifiées in vivo à ce jour et il permet enfin de comprendre comment les protéines avec MYR sont distribuées dans différents compartiments subcellulaires. Enfin, ces études ont révélé que MYR implique un ensemble non négligeable de cibles chevauchantes possédant une autre modification N-terminale largement répandue, la N-alpha-acétylation. Ceci pose la question de la fonction cellulaire de protéoformes qui peuvent subir deux modifications distinctes. Enfin, les auteurs ont identifié les empreintes signature de l’ajout d’un autre acide gras, le palmitate. Ils ont découvert que cette PM est imprimé sur les gènes codant certaines protéines possédant MYR, ce qui permet la reconnaissance visuelle facile des séquences présentant les deux modifications. L’identification de la MYR que l’étude présente résout donc l’identification de la seconde modification.
Dans l'ensemble, ces études récentes permettent (i) d'accéder à la face largement cachée de l'une des modifications protéiques les plus intrigantes des organismes pluricellulaires, (ii) d'identifier plus d'un millier de nouvelles protéines la portant et (iii) en révéler le caractère hétérogène dans les deux organismes étudiés.
© Thierry Meinnel & Carmela Giglione
En savoir plus
-
Castrec B, Dian C, Ciccone S, Ebert CL, Bienvenut WV, Le Caer JP, Steyaert JM, Giglione C, Meinnel T.
Nature Chemical Biology 2018 14: 671-679 doi: 10.1038/s41589-018-0077-5. Epub 2018 Jun 11 -
Majeran W, Le Caer JP, Ponnala L, Meinnel T, Giglione C.
Plant Cell. 2018 Mar;30(3):543-562. doi: 10.1105/tpc.17.00523. Epub 2018 Feb 16.
-
Lipid Anchor: Postal Code for Proteins on the Road to Membranes