Mécanisme moléculaire de prédation par contact chez une bactérie prédatrice
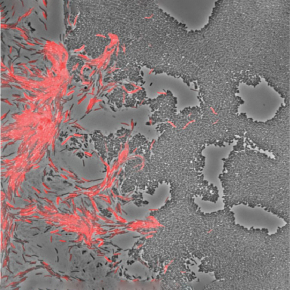
Les bactéries du sol ont la capacité de se déplacer et d’attaquer collectivement d’autres microorganismes pour s’en nourrir. Les mécanismes moléculaires permettant cette prédation sont restés longtemps inconnus. Les scientifiques montrent que la bactérie Myxococcus xanthus tue sa proie par contact direct, ce qui résulte de l’activité d’un nouveau type de complexe protéique transmembranaire, baptisé « Kil ». Le système Kil est dynamiquement assemblé au contact de la proie, stoppant le mouvement de la bactérie prédatrice et entrainant la lyse de la proie en quelques minutes. Ce travail, publié dans la revue Elife, pose donc les premières bases moléculaires d’un de prédation potentiellement conservé chez les bactéries prédatrices.
Myxococcus xanthus est une bactérie environnementale présente dans le sol. Elle est dite « sociale » car capable de se déplacer en groupe de manière coordonnée, semblable aux comportements collectifs chez les animaux. Cette bactérie «attaque» collectivement d’autres microorganismes (bactéries, champignons microscopiques etc..) pour s’en nourrir. Il a été longtemps considéré que M. xanthus tue ses proies par des mécanismes divers, en secrétant des molécules lytiques, protéases, lipases, antibiotiques, lysant ainsi les cellules proies dans le milieu extracellulaire pour en assimiler les nutriments.
Grâce à des techniques d’imagerie nouvelles, les scientifiques ont observé que le processus de prédation est initié par des bactéries motiles qui, à la manière d’éclaireurs, pénètrent une colonie proie et initient l’invasion ultérieure par de plus larges groupes. Remarquablement, la pénétration des éclaireurs est rendue possible par leur capacité à tuer les bactéries proies par contact, un processus au cours duquel les cellules prédatrices s’immobilisent le temps de lyser la proie.
Les chercheurs ont donc cherché à comprendre les mécanismes moléculaires permettant la perception de la proie et la lyse cellulaire. Ils ont ainsi identifié un nouveau système moléculaire essentiel aux deux phénomènes, baptisé "Kil". Kil présente de fortes similitudes avec un pilus de type Tad (Tight adherence), impliqué, chez de nombreuses bactéries, dans la régulation de la formation de biofilms. En marquant par fluorescence certaines protéines Kil, les scientifiques ont directement observé que le système Kil s’assemble au point de contact avec la proie, provoquant la rupture locale de son enveloppe cellulaire. L’inactivation du système Kil rend les bactéries prédatrices « aveugles » à leur proie et incapables de les tuer. Le système Kil est donc indispensable à la nutrition de M. xanthus. Cette découverte pose donc les bases moléculaires du processus de prédation chez M. xanthus, mais aussi chez d’autres bactéries prédatrices (comme Bdellovibrio ou Bacteriovorus) dans le génome desquelles des homologues du système Kil semblent présents.
De nombreuses questions restent en suspens: Par quel mécanisme l’assemblage du système Kil signale-il l’arrêt de la motilité? Quelle est l’identité du système d’intoxication et comment Myxococcus fait-il la distinction entre elle-même et une proie ? Existe-t-il des mécanismes d’immunité ? Autant de questions auxquelles les chercheurs s'attacheront à répondre dans le futur.
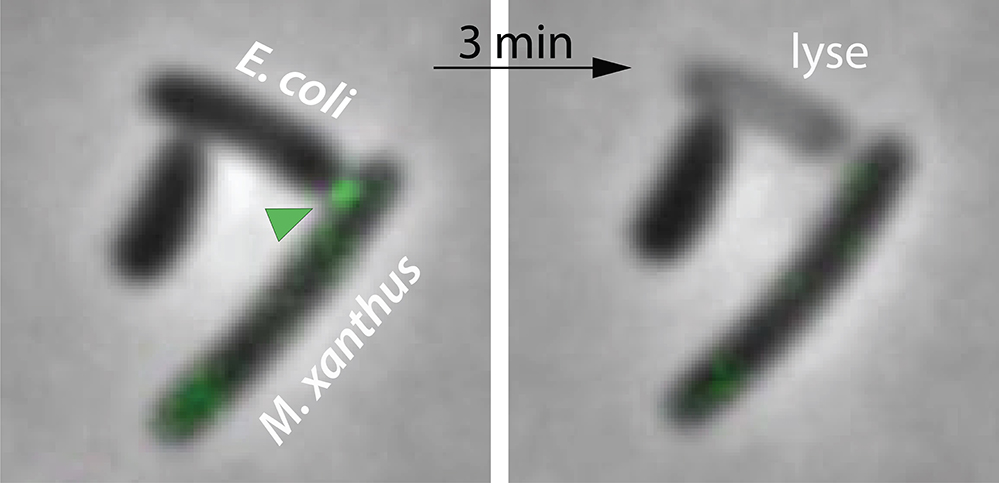
Figure : A droite : une cellule de M. xanthus est entrée en contact avec une cellule d’ Escherichia coli (proie). On peut observer le système Kil marqué par l’expression d’une protéine fluorescente verte (flèche verte) assemblé au point de contact entre les deux bactéries. A gauche : au bout de quelques minutes, la cellule proie est lysée ce qui est détecté par une perte de contraste. Le système Kil est alors désassemblé et la motilité de M. xanthus est ré-initiée, permettant l’interaction avec de nouvelles proies.
Pour en savoir plus :
A Tad-like apparatus is required for contact-dependent prey killing in predatory social bacteria. Seef S, Herrou J, de Boissier P, My L, Brasseur G, Robert D, Jain R, Mercier R, Cascales E, Habermann BH, Mignot T.
Elife. 2021 Sep 10. doi: 10.7554/eLife.72409.
Contact
laboratoire
Laboratoire de chimie bactérienne (CNRS/Aix-Marseille Université)
31 Chemin Joseph Aiguier
Marseille cedex 13420 - France