L’uridylation empêche la production de petits ARNs délétères chez Arabidopsis
Les ARN messagers (ARNm) des plantes doivent être protégés d’un mécanisme de défense contre les virus sous peine d’autodestruction. Les plantes produisent en effet des petits ARN complémentaires aux ARN viraux à détruire mais ce mécanisme ne doit pas s’attaquer aux ARNm endogènes. Dans une étude publiée dans la revue Nature Communications, les chercheurs ont montré que l’ajout d’uridines empêche les ARNm endogènes de devenir une source de petits ARN délétères pour le développement et la survie chez la plante modèle Arabidopsis thaliana.
La dégradation des ARN est essentielle à la régulation de l’expression du génome mais également à la lutte contre les virus. Chez les plantes, la reconnaissance d’ARN exogènes comme les ARN viraux déclenche la production de petits ARN complémentaires induisant leur destruction. Ce mécanisme de restriction virale est strictement contrôlé dans la cellule afin de le limiter aux ARN exogènes (et à quelques ARN endogènes). Il est essentiel pour la survie de la plante que ses propres ARN messagers (ARNm) échappent à ce système de surveillance. En effet, des intermédiaires de dégradation des ARNm endogènes reconnus comme aberrants par la machinerie cellulaire peuvent devenir une source de petits ARN délétères. En cas de production, ces petits ARN entrainent une dégradation anarchique des ARNm, compromettant le développement et la survie de la plante.
Pourtant, si une dégradation incontrôlée des ARNm est néfaste, chaque ARNm doit être éliminé de manière régulée afin d’assurer le développement de la plante et son adaptation aux changements environnementaux. La dégradation contrôlée des ARNm est initiée soit par un clivage endoribonucléolytique, soit par l’élimination des protections présentes aux extrémités des ARNm : un nucléotide modifié appelé coiffe à l'extrémité 5'et une succession d’adénosines appelée queue poly(A) en 3'. Ces intermédiaires de dégradation doivent impérativement être dégradés efficacement pour éviter d’être reconnus par la machinerie de surveillance et induire la production de petits ARN délétères.
Les chercheurs avaient précédemment montré que le raccourcissement de la queue poly(A) dans la plante modèle Arabidopsis thaliana est suivi de l’ajout de quelques uridines par une enzyme appelée UTP:RNA URIDYLYLTRANSFERASE 1 (URT1). Dans ce nouveau travail, ils montrent que URT1 participe à un réseau moléculaire connectant des facteurs impliqués dans la dégradation des ARNm. En particulier, URT1 interagit directement avec DECAPPING5 (DCP5), un facteur activant l’enlèvement de la coiffe des ARNm. Cette interaction est inédite chez les eucaryotes et participe à expliquer comment l’uridylation promeut la dégradation des ARNm chez Arabidopsis.
De plus, grâce à l’obtention de données de séquençage direct de l’ARN par nanopores, les chercheurs montrent que URT1 a également un rôle global dans le contrôle de la taille des queues poly(A) et prévient l’accumulation d’ARNm avec des queues poly(A) trop raccourcies. Ainsi, en favorisant l’élimination d’intermédiaires de dégradation des ARNm et en empêchant l’accumulation d’ARNm « aberrants », l’uridylation par URT1 limite la production de petits ARNs néfastes pour la plante. Cela met en lumière des fonctions insoupçonnées de l’uridylation dans le métabolisme des ARNm chez les plantes.
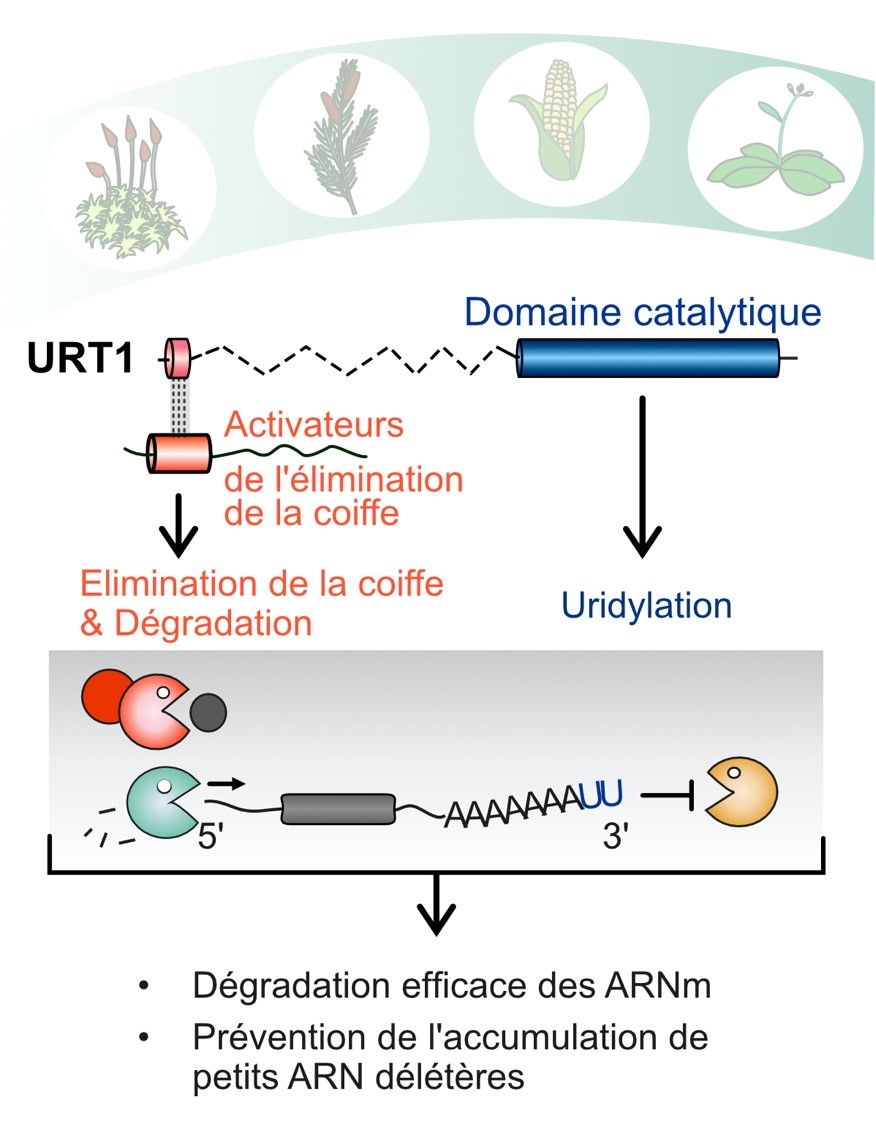
Figure : L’uridylation permet une dégradation efficace des ARNm et les empêche de devenir une source de petits ARN délétères chez Arabidopsis. UTP:RNA URIDYLYLTRANSFERASE 1 (URT1) interagit directement avec un facteur activant l’enlèvement de la coiffe des ARNm via un motif conservé chez les plantes. URT1 prévient l’accumulation d’ARNm avec des queues poly(A) excessivement raccourcies.
Pour en savoir plus
The TUTase URT1 connects decapping activators and prevents the accumulation of excessively deadenylated mRNAs to avoid siRNA biogenesis.
Scheer H, de Almeida C, Ferrier E, Simonnot Q, Poirier L, Pflieger D, Sement FM, Koechler S, Piermaria C, Krawczyk P, Mroczek S, Chicher J, Kuhn L, Dziembowski A, Hammann P, Zuber H, Gagliardi D.Nat Commun. 2021 Feb 26;12(1):1298. doi: 10.1038/s41467-021-21382-2.
Contact
Laboratoire
Institut de biologie moléculaire des plantes (IBMP) - (CNRS / Université de Strasbourg)
12, rue du général Zimmer
67084 Strasbourg
