Les génomes de centaines d’espèces éteintes reconstruits automatiquement
Une publication dans la revue Nature Ecology and Evolution décrit comment les scientifiques ont développé un nouvel algorithme appelé AGORA pour explorer l’évolution des génomes. Nourri de centaines de génomes modernes, AGORA a reconstruit automatiquement la structure partielle de celui de leurs ancêtres. Ce travail établit un cadre robuste pour comprendre l’évolution des génomes.
Nous l’oublions parfois, mais la biologie est une science dite « historique », car elle s’appuie sur l’histoire du vivant. Ainsi, les génomes d’espèces ancestrales sont des témoins du passé des organismes, au même titre que les fossiles, et ils sont essentiels pour retracer les changements qui ont eu lieu au cours de l’évolution, que ce soit au niveau des mutations à petite échelle aussi bien qu’au niveau des génomes ou des espèces. Bien qu’il soit possible aujourd’hui de séquencer directement l’ADN d’organismes partiellement préservés dans des conditions particulières, comme le permafrost, seuls les organismes qui vivaient il y a moins d’un million d’année sont accessibles par cette approche. Par comparaison, l’ancêtre des humains et des chimpanzés vivait il y a plus de 6 millions d’années. Ainsi, pour remonter plus loin dans le temps, il est nécessaire de recourir à des reconstructions par des approches informatiques, à partir de génomes modernes. Cette discipline est encore en émergence, et les reconstructions sont peu nombreuses et difficiles à utiliser, ce qui limite les possibilités de réaliser des analyses à grande échelle pour obtenir une vue précise et globale de l’évolution de l’architecture des chromosomes et des génomes au cours de l’évolution.
Un grand pas en avant dans ce domaine a été franchi grâce à un nouvel algorithme appelé AGORA, pour Algorithm for Gene Order Reconstruction in Ancestors. Cette méthode s’appuie avant tout sur les génomes d’espèces modernes, dont des centaines sont aujourd’hui disponibles dans les bases de données. Les scientifiques utilisent d’abord la séquence des gènes identifiés dans ces génomes pour reconstruire des familles de gènes qui descendent d’un même gène ancestral apparu à un moment donné de l’évolution. Chaque famille peut compter des milliers de gènes, qui sont répartis dans des centaines de génomes appartenant à des espèces couvrant des centaines de millions d’années d’évolution. Par exemple, un gène apparu chez l’ancêtre des vertébrés sera aujourd’hui encore représenté par des versions modernes chez les poissons, les oiseaux et les mammifères. Chaque version est légèrement différente des autres par sa séquence d’ADN à cause des mutations qui se sont produite au cours du temps, mais elles sont néanmoins reconnaissables par des logiciels de comparaison de séquences. L’étape suivante consiste à reconstruire l’histoire évolutive de chaque famille, grâce à des logiciels s’appuyant sur un formalisme mathématique aujourd’hui bien maîtrisé. C’est ensuite qu’AGORA entre en jeu. Grâce à cette histoire évolutive et à la position des gènes modernes dans leurs génomes respectifs, l’ordre des gènes le long des chromosomes des espèces ancestrales est déduit à partir de celui des espèces modernes, chez qui il est partiellement préservé. L’algorithme s’appuie sur une structure en forme de graphe qui est parcouru de manière très efficace afin de déterminer quel gène est voisin de quel autre gène dans le génome ancestral.
Les résultats publiés dans la revue Nature Ecology and Evolution décrivent comment AGORA a ainsi pu reconstruire la structure partielle de centaines de génomes ancestraux, une première. Les scientifiques s’en sont ensuite servis pour calculer la fréquence des réarrangements de chromosomes avec une précision inégalée. Cette ressource permet également d’avoir rapidement accès aux régions des génomes modernes qui ont la même structure que celle d’ancêtres extrêmement anciens. Si ces régions restent inchangées, c’est la signature probable d’une grande importance biologique. Ces travaux établissent donc un cadre solide pour l’étude de l’évolution de la structure des génomes, et ce faisant permettent de mieux prendre en compte la dimension « historique » de la biologie.
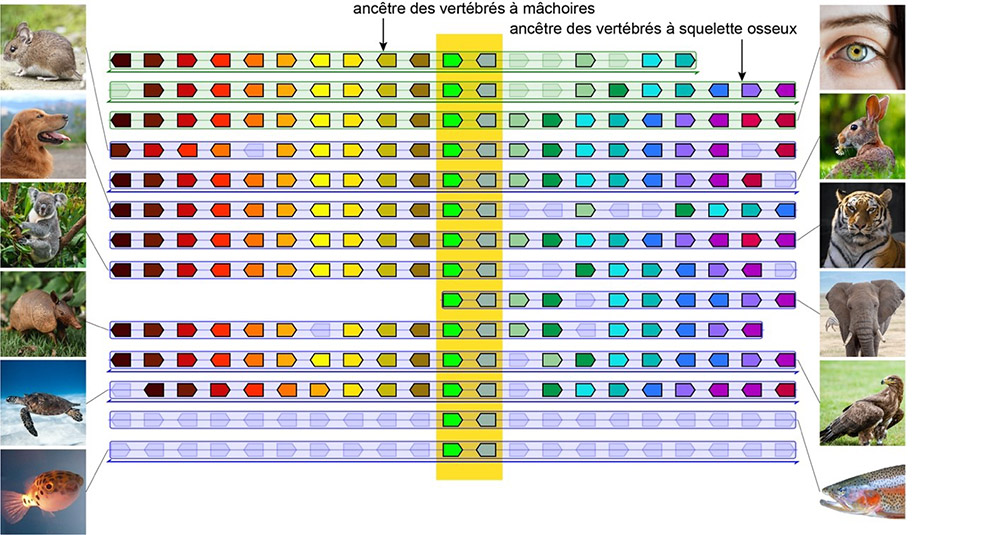
Figure : Le panneau central représente des fragments de génomes d’espèces de vertébrés modernes. Chaque flèche représente un gène, dont la couleur dénote l’appartenance à une même famille. Les deux fragments de génomes en haut sont des reconstructions inférées par AGORA pour l’ancêtre des vertébrés à mâchoires (Gnathostomes) et des vertébrés à squelette osseux (Euteleostomi). Les deux gènes (surlignée en jaune), ELK3 et CDK17, sont voisins dans tous les génomes de vertébrés séquencés, et à ce titre représentent la paire de gènes dont l’adjacence est la plus conservée dans ce groupe au cours de l’évolution, probablement en raison de contraintes biologiques fortes.
Pour en savoir plus :
Reconstruction of hundreds of reference ancestral genomes across the eukaryotic kingdom.
Muffato, M., Louis, A., Nguyen, N.T.T. et al.
Nat Ecol Evol (2023). DOI:https://doi.org/10.1038/s41559-022-01956-z
Contact
Laboratoire
Institut de biologie de l’Ecole normale supérieure - IBENS (CNRS/ENS/Inserm)
46 rue d’Ulm
75005 Paris
