Le talon d’Achille des bactéries pathogènes : le mécanisme de formation de leur paroi
Les phénomènes de résistance aux antibiotiques, développés par plusieurs pathogènes bactériens, sont de plus en plus répandus. Ceci souligne l’importance d’une recherche visant à comprendre les processus importants pour la survie bactérienne. En s'appuyant sur la cryo-microscopie électronique, la diffraction aux rayons-X, et des méthodes biophysiques et de génétique bactérienne, les scientifiques ont caractérisé des formes auto-assemblées de la protéine MreC, essentielle pour l’élongation de la paroi bactérienne. Ces résultats sont publiés dans la revue Nature Communications.
La paroi cellulaire joue un rôle primordial dans la survie des bactéries, et depuis des décennies le mécanisme de formation de son composant central, le peptidoglycane, est exploité en tant que cible des antibiotiques de type beta-lactamine, comme les pénicillines et céphalosporines. Le peptidoglycane forme une structure qui ressemble à un ‘filet de pêcheur’ et qui entoure toute la bactérie. Il est essentiel non seulement pour sa stabilité mais aussi pour le bon déroulement des différentes étapes du cycle cellulaire, comme la division et l’élongation de la paroi du microorganisme. Les protéines qui participent à la machinerie de synthèse de ce ‘filet’ sont indispensables pour la survie bactérienne, et malgré les phénomènes de résistance aux antibiotiques qui sont de plus en plus répandus dans les hôpitaux et cliniques du monde entier, le processus de formation de la paroi des bactéries continue à être une cible de choix pour un éventuel développement de nouveaux agents antibactériens.
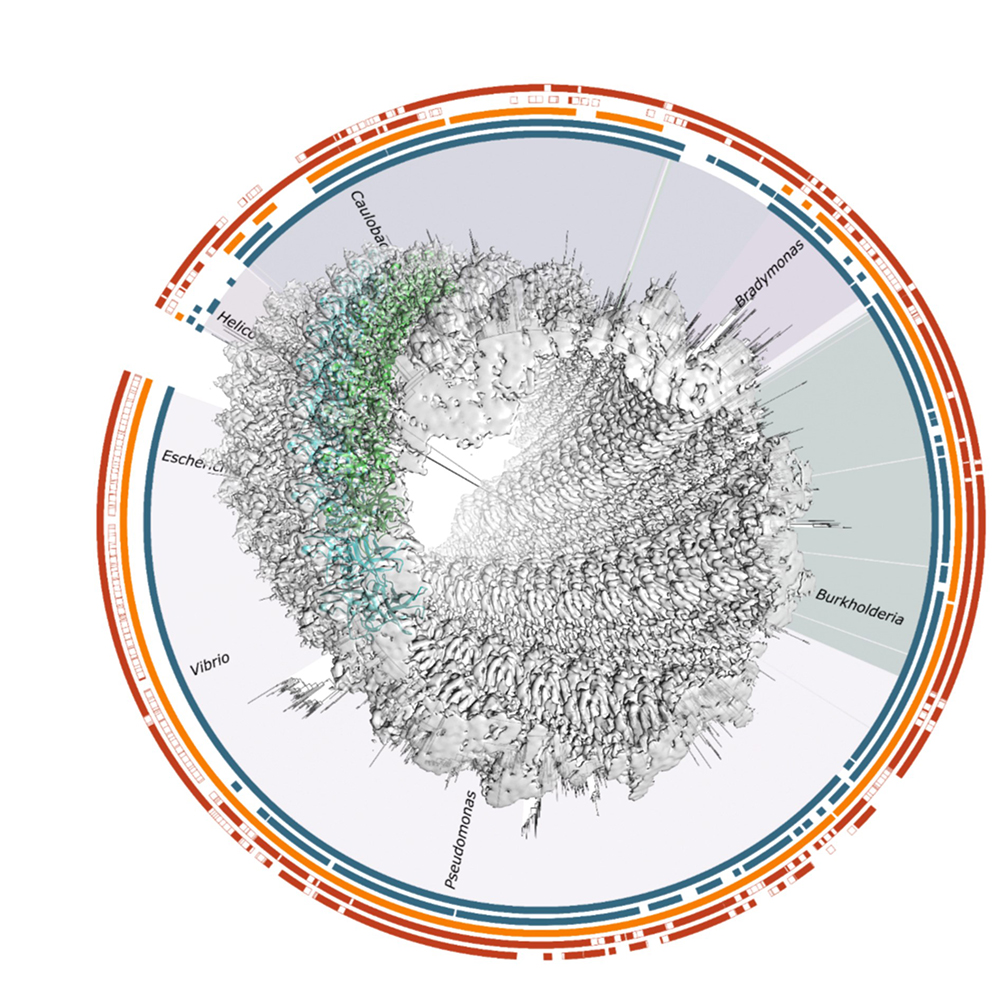
Une des protéines qui joue un rôle central dans la formation de la paroi bactérienne est MreC. Cette protéine est supposée former une plateforme permettant la stabilisation d’autres protéines qui participent au processus d’élongation de la cellule. Ce travail montre comment MreC est capable de s’auto-associer et s’organiser sous la forme de différents polymères comme des filaments et même de tubes. Les scientifiques ont étudié MreC de trois pathogènes différents : Escherichia coli, Acinetobacter baumannii, et Pseudomonas aeruginosa. La protéine de cette dernière bactérie, un pathogène nosocomial qui cause des infections graves chez l’homme, a été choisie pour les études structurales. Les structures obtenues à partir d’images de cryo-microscopie électronique (données collectées au microscope Glacios à l’IBS) ainsi que par cristallographie aux rayons X (données à résolution atomique collectées au synchrotron LNLS à Campinas, Brésil), ont révélé des régions clé pour la formation des formes tubulaires de MreC. Toute perturbation de cette forme polymérisée, par exemple par l’introduction de mutations, a impacté non seulement la capacité de MreC à produire des polymères, mais également la production de MreC elle-même dans la cellule bactérienne, un phénomène vérifié par des expériences effectuées directement dans des souches de P. aeruginosa. Les surfaces d'interaction entre les différents éléments constitutifs des polymères de MreC pourraient être une cible potentielle pour le développement d'inhibiteurs totalement novateurs.
Pour en savoir plus :
Self-association of MreC as a regulatory signal in bacterial cell wall elongation.
Martins A, Contreras-Martel C, Janet-Maitre M, Miyachiro MM, Estrozi LF, Trindade DM, Malospirito CC, Rodrigues-Costa F, Imbert L, Job V, Schoehn G, Attrée I, and Dessen A
Nature Communications 20 mai 2021. https://doi.org/10.1038/s41467-021-22957-9
Contact
laboratoire
Institut de Biologie Structurale (CNRS, CEA, Univ. Grenoble Alpes)
71 avenue des Martyrs
38000 Grenoble