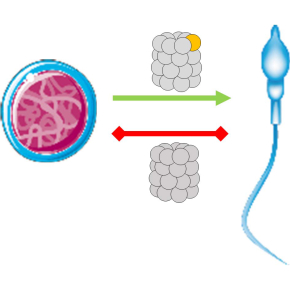
Le spermatoprotéasome : un complexe enzymatique clé de la spermatogénèse
Le spermatoprotéasome est trouvé exclusivement dans les cellules germinales des mammifères et est indispensable à la spermatogénèse. Il diffère du protéasome constitutif par la présence d’une seule protéine alternative sur les 14 constituant le complexe. Ce travail, publié dans la revue PNAS, montre que les quelques acides aminés qui particularisent le spermatoprotéasome sont suffisants pour induire une plus forte dynamique structurale et ainsi changer son interactome et donc sa fonction.
Le protéasome permet de dégrader les protéines intracellulaires non fonctionnelles afin d’éviter leur agrégation qui pourrait être délétère, mais aussi de réguler la concentration de certaines protéines clés. A ce titre, le protéasome est au cœur de nombreuses voies de signalisation cellulaires. Sa localisation intracellulaire, encore mal décrite semble également cruciale pour réguler les acteurs de certains processus cellulaires (réplication, traduction, différentiation…).
Au cours de l’évolution, le protéasome s’est diversifié afin d’assumer différents rôles, en fonction de l’environnement cellulaire. Différents types de protéasomes, contenant des sous-unités alternatives, sont ainsi apparus, tel que le spermatoprotéasome (s20S) dans les cellules germinales (ovaires et testicules). Il diffère du protéasome constitutif (c20S) par sa sous-unité non-catalytique α4s qui remplace la sous-unité constitutive α4.
Le rôle crucial de cette sous-unité α4s dans la spermatogénèse a été démontrée chez la souris : les cellules germinales n’exprimant pas cette protéine bloquent leur différenciation. Cependant, les raisons pour lesquelles le remplacement d’une seule sous-unité dans le protéasome 20S est indispensable à ce processus restent non déterminées. Pour mieux comprendre les relations structure-fonction du s20S pendant la spermatogénèse, les scientifiques ont analysé des cellules germinales de mammifères à différents états de différenciation avec des stratégies avancées de spectrométrie de masse. Ils ont observé une incorporation brusque d’α4s (>98%) au cours de la spermatogénèse et une très forte activation concomitante du s20S. De plus, le s20S, contrairement au c20S, interagit avec des protéines du complexe synaptonémal, crucial pour la recombinaison homologue des chromosomes lors de la meïose. Enfin, une approche structurale a permis de découvrir qu’α4s est plus dynamique en solution qu’α4, rationnalisant ainsi les bases moléculaires de la spécificité fonctionnelle de ce sous-type de protéasome. Ces résultats pourraient permettre de mieux comprendre les mécanismes d’infertilité masculine.
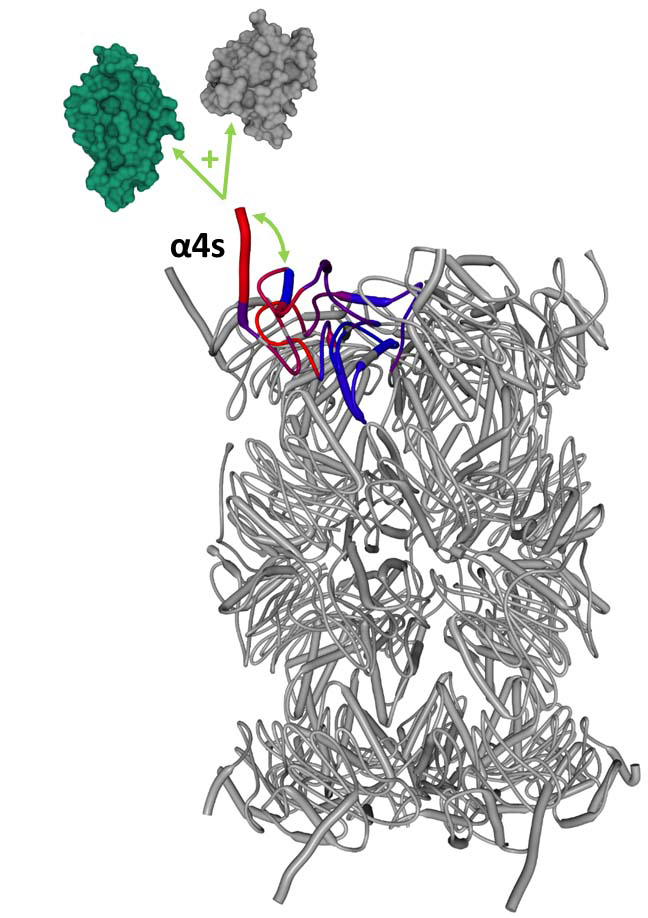
Figure : Modèle du spermatoprotéasome avec la sous-unité α4s en fonction de la dynamique et de l'exposition au solvant de ses acides aminés (bleu : faible, rouge : fort). Une dynamique plus forte de l’extrémité C-terminale induit des différences dans l’interactome du protéasome et donc dans sa fonction et localisation cellulaire.
Pour en savoir plus :
Proteasome complexes experience profound structural and functional rearrangements throughout mammalian spermatogenesis.
Živković D, Sanchez Dafun A, Menneteau T, Schahl A, Lise S, Kervarrec C, Toste Rêgo A, da Fonseca PCA, Chavent M, Pineau C, Burlet-Schiltz O, Marcoux J, Bousquet MP.
Proc Natl Acad Sci U S A. 4 avril 2022. doi: 10.1073/pnas.2116826119.
Contact
Laboratoire
Institut de pharmacologie et de biologie structurale (CNRS/Université Paul Sabatier)
BP64182
205 route de Narbonne
31077 Toulouse Cedex04