Le chaudron évolutif des Nidovirales
Les virus à ARN positifs sont contraints de protéger leur génome. Pour ce faire, ils utilisent des enzymes qui vont modifier l'ARN afin d'assurer la stabilité et l’échappement au système immunitaire. Dans cet article, publié dans la revue Nucleic Acids Research, les scientifiques caractérisent une de ces enzymes dans une famille de virus parente des Coronavirus. L'émergence de cette dernière montre que la fonction est critique et que son évolution n'est pas linéaire.
Les Nidovirales constituent un ordre viral qui regroupe divers familles de virus, notamment les Coronaviridae, dont la particularité est de posséder des génomes à ARN de grande taille. D'une manière générale, l'organisation génomique des Nidovirales présente deux grands cadres de lecture ouverts (ORF1a et ORF1b). L'ORF1a code pour les protéines d'échafaudage du complexe de réplication (activateur de protéines, et enzymes antagonistes du système immunitaire inné) tandis que l'ORF1b code pour les enzymes de synthèse, d'édition ou de dégradation de l'ARN, y compris toutes les méthyltransférases actuellement connues. Pour la famille des Coronaviridae (CoV), les méthyltransférases N7 et 2'O de la coiffe protégeant l’ARN de la dégradation ont été bien caractérisées, résidant respectivement dans nsp14 et nsp16, tous deux dans l'ORF1b.
Dans le cas des Tobaniviridae, autre famille des Nidovirales, seule la 2'O-méthyltransférase, située à l'extrémité de l'ORF1b, a pu être facilement identifiée, tandis que la présence probable d'une N7-méthyltransférase dans l'ORF1a, une localisation étrange pour ce gène, restait discutée. Dans cet article, les scientifiques démontrent que ce domaine abrite effectivement une activité N7-méthyltransférase spécifique, ce qui rompt avec le concept commun selon lequel Orf1b porte ce type d'enzymes d'édition/réplication de l'ARN. Ils présentent sa structure à très haute résolution, résolue par cristallographie aux rayons X et l’utilisation d’un modèle structurel distant généré par Alphafold2. Cette structure montre une MTase à repliement canonique, malgré une conservation de séquence très limitée avec les N7-MTases connues. Cette MTase ORF1a de nidovirus est donc remarquablement différente de la nsp14 des CoVs.
Cette nouvelle structure et cette activité sont essentielles pour comprendre les différentes stratégies développées par les nidovirus pour coiffer leur ARN génomique. Elle souligne à quel point cette fonction de méthylation est critique pour la survie de ces virus, et montre également que dans cet ordre, différents événements d'acquisition de gènes ont eu lieu, posant la question des avantages évolutifs d'une telle apparition à cet endroit inattendu du génome chez les virus infectant les animaux à sang froid. Enfin, ce virus en apparence insignifiant, comme d'autres virus mal connus, pourrait fournir des outils biotechnologiques pour la stabilisation des ARN thérapeutiques comme les vaccins ARNm.
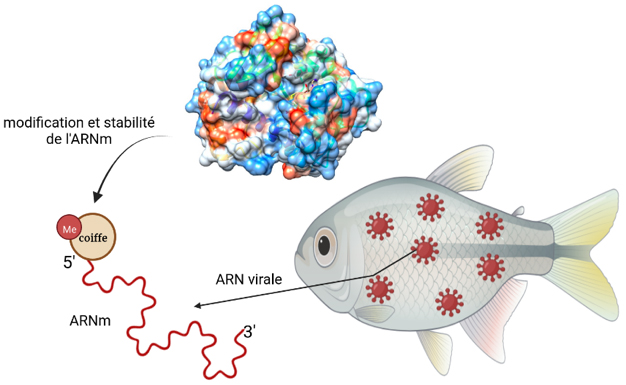
Figure : Le génome des Tobanivirus encode une N7 méthyltransférase spécifique qui vise à protéger l'ARN génomique et à éviter la réponse immunitaire innée. Cette structure est la première de ce type décrite chez un virus à ARN à brin positif.
Pour en savoir plus :
A second type of N7-guanine RNA cap methyltransferase in an unusual locus of a large RNA virus genome
Ashleigh Shannon, Bhawna Sama, Pierre Gauffre, Théo Guez, Françoise Debart, Jean-Jacques Vasseur, Etienne Decroly, Bruno Canard and François Ferron
Nucleic Acids Research 21 octobre 2022. https://doi.org/10.1093/nar/gkac876
Contact
Laboratoire
Architecture et fonction des macromolécules biologiques (CNRS/Aix-Marseille Université)
163 avenue de Luminy
13009 Marseille
