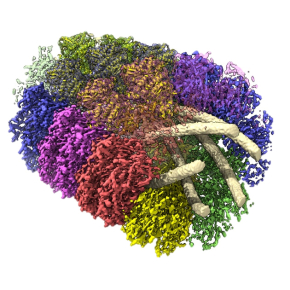
La structure du chromosome de mimivirus à 3.7 Å révèle un paradoxe évolutif
Mimivirus est le premier virus géant de la famille des Mimiviridae. Avec un génome ADN de 1.2 million de paires de bases codant pour ~1000 protéines, mimivirus a révolutionné la perception traditionnelle des virus et inspiré de nouvelles hypothèses sur leur évolution en lien avec celle du monde cellulaire. Ces travaux, publiés dans la revue eLife, présentent l’organisation du génome de mimivirus au sein de la particule virale. Cette structure est étonnamment constituée par l’empilement hélicoïdal d’une enzyme (une oxydoréductase) sans lien fonctionnel connu avec l’ADN.
Depuis sa découverte en 2003, mimivirus a été le plus étudié des virus géants infectant les amibes du genre Acanthamoeba. Ces travaux ont porté sur l’imagerie de son cycle réplicatif, la fonction de ses gènes et l’enchaînement de leur expression (transcriptome), sa composition protéique (protéome), puis sur la détermination détaillée de la structure tridimensionnelle de sa particule virale icosaédrique aux dimensions spectaculaires (0.75 micron de diamètre), recouverte d’une couche de fibres lui donnant une apparence « chevelue » caractéristique en microscopie électronique. Mais nous ne savions rien de la façon dont son génome pouvait se compacter au centre de la particule virale (le nucléoïde), ce dernier apparaissant comme une simple boule noire (dense aux électrons) d’environ 350 nm au centre de la particule.
Lors de travaux initiés en 2007, les scientifiques ont d’abord dû mettre au point un protocole d’ouverture des capsides virales suffisamment doux pour en extraire le nucléoïde en en préservant la structure. C’est alors qu’ils ont pu découvrir des fibres longues de plusieurs microns et d’environ 30 nm de diamètre.
La structure de ces fibres a été analysées par cryo-microscopie électronique pour en obtenir le détail à une résolution de 3.7 Å montrant l’enchevêtrement d’une hélice à 5 brins constituée de protéines formant une coquille externe de 8 nm d’épaisseur, bordée de 5 brins de double-hélice ADN et contenant l’ARN polymérase en son centre. Une conformation alternative de la fibre constituée d’une hélice à 6 brins imbriqués d’une manière similaire a également été identifiée, à 4 Å de résolution.
Au-delà de la structure originale de ce chromosome viral, une autre surprise attendait les chercheurs. Si les analyses protéomiques préalables des fibres purifiées avaient suggéré la prépondérance de deux oxydoréductases virales, la structure obtenue par microscopie électronique a permis de confirmer sans ambiguïté qu’elles constituaient bien l’échafaudage des hélices protéiques.
La découverte est inattendue pour deux raisons ; La première est que ce type de protéines (des enzymes assez banales du métabolisme), n’a jamais été impliqué dans la compaction des acides nucléiques ; La seconde est que ces mêmes protéines étaient déjà connues pour composer les « cheveux » qui décorent la particule de mimivirus.
L’usage parcimonieux du même couple de protéines dans deux compartiments différents sans la moindre relation fonctionnelle est particulièrement paradoxal et interroge sur les pressions évolutives conduisant à une telle organisation pour un virus codant pour près d'un millier de protéines.
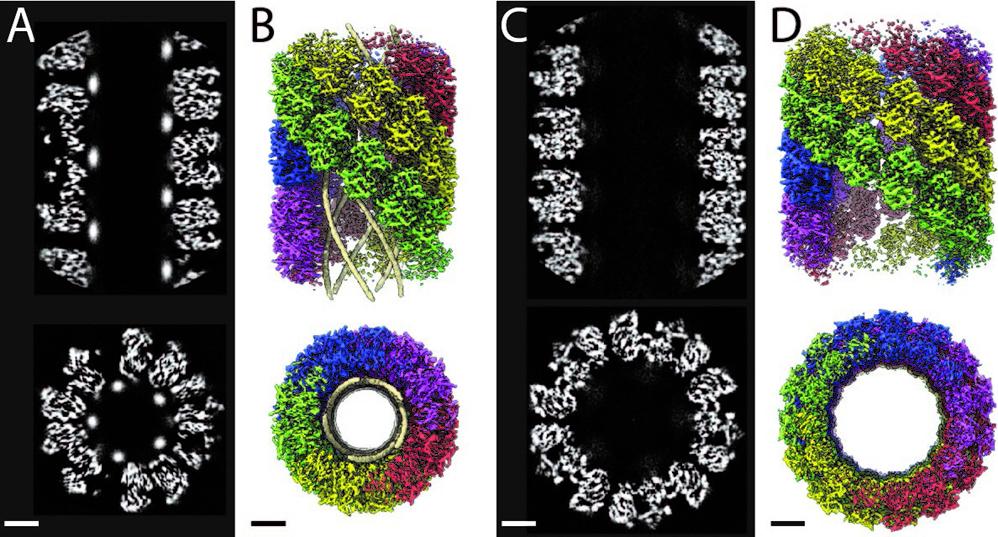
Figure : Cartes de microscopie électronique de la fibre génomique de mimivirus. A, C : Coupes longitudinales (haut) et transversales (bas) au milieu de la carte de la forme compacte (A) contenant l’ADN et de la forme relaxée (C) où l’ADN n’est plus visible. B,D : Vue longitudinale (haut) et orthogonale (bas) des cartes de microscopie électroniques avec chaque brin du ruban formant l’hélice coloré différemment pour la forme compacte (B) et la forme relaxée (D). Quelques unités répétées en surface de la carte ont été supprimées pour permettre de voir les brins d’ADN dans la forme compacte. Barre d’échelle 5 nm.
Pour en savoir plus :
The giant mimivirus 1.2 Mb genome is elegantly organized into a 30 nm diameter helical protein shield.
Villalta A, Schmitt A, Estrozi LF, Quemin ERJ, Alempic JM, Lartigue A, Pražák V, Belmudes L, Vasishtan D, Colmant AMG, Honoré FA, Couté Y, Grünewald K, Abergel C.
eLife. 28 juillet 2022 doi: 10.7554/eLife.77607.
Contact
Laboratoire
Laboratoire information génomique et structurale (CNRS/Université Aix-Marseille)
163 Avenue de Luminy
Case 934
13288 Marseille cedex 09