La régulation des étiquettes moléculaires sur les ARN du VIH : une nouvelle piste antivirale identifiée
L’infection par le Virus de l'Immunodéficience Humaine (VIH-1) débute par la copie de son génome viral ARN en ADN. Des antiviraux utilisés en thérapie inhibent cette étape, et bloquent l'infection. Dans une étude publiée dans la revue Nucleic Acids Research, des scientifiques ont démontré que de petites étiquettes moléculaires, appelées 2’-O-méthylations, qui décorent le génome ARN du VIH-1, inhibent la copie du génome viral ARN en ADN, limitant ainsi l'infection des cellules dormantes (les cellules qui ne se divisent pas). Cette découverte ouvre de nouvelles perspectives sur le contrôle de l'infection au VIH-1.
Le virus de l’immunodéficience humaine de type 1 (VIH-1) représente un problème de santé public important, et le contrôle de la réplication du virus par des molécules antivirales est un enjeu majeur. L’infection des cellules par le virus requiert une étape de transcription inverse efficace : c’est la copie du génome ARN du virus en ADN, qui est intégré au génome ADN des cellules de l’hôte humain.
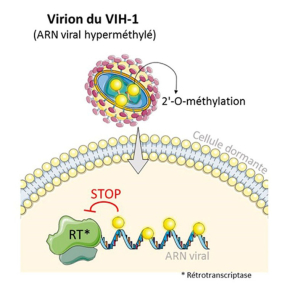
Dans un travail publié dans la revue Nucleic Acids Research, des scientifiques ont décrypté un nouveau mécanisme de régulation de l’étape de transcription inverse. En effet, il avait été précédemment montré que le génome ARN du VIH-1 est décoré par de petites étiquettes moléculaires (aussi appelées marques épitranscriptomiques), telles que les 2′-O-méthylations, mais leur effet sur l’étape de transcription inverse n’était pas connu.
Cette étude montre que, lors de l’infection, la protéine virale capable d’effectuer la transcription inverse (appelée RT) s’arrête au niveau de ces 2′-O-méthylations. Cela limite la copie complète du génome du virus. L’arrêt de la copie cible principalement l’infection de cellules qui ne se divisent pas (appelées cellules quiescentes, ou dormantes), car ces cellules ont une faible quantité de nucléotides (petites molécules nécessaires à fabrication d’ADN). Les scientifiques montrent ainsi que l’accumulation d’étiquettes moléculaires, ici les 2′-O-méthylations, sur le génome du virus HVI-1, a un rôle antiviral.
Une régulation fine entre effets pro-viraux et antiviraux
De façon contradictoire, des travaux antérieurs avaient montré que ces mêmes modifications avaient des effets pro-viraux sur la réplication du VIH : elles protègent le génome ARN du virus de la détection par des senseurs intracellulaires de l’immunité innée (comme MDA5) d’une part, et d’autre part, elles limitent la dégradation du génome ARN par une molécule appelée ISG20.
Cette nouvelle étude suggère donc l’existence d’une fine régulation de la quantité de 2′-O-méthylations sur le génome ARN viral, qui favoriserait à la fois l’échappement à la détection par MDA5 et la dégradation par ISG20, sans toutefois empêcher totalement la transcription inverse, car la copie entière du génome est nécessaire à l’infection productive des cellules.
Ces résultats, permettront d’initier des projets de développement de nouvelles stratégies visant à cibler le niveau de méthylation des ARN viraux : si ce niveau est diminué, cela stimule la capacité naturelle des cellules à détecter et dégrader les ARN viraux, tandis que si ce niveau est augmenté, cela limite la réplication virale et notamment la transcription inverse dans le cas du VIH-1.
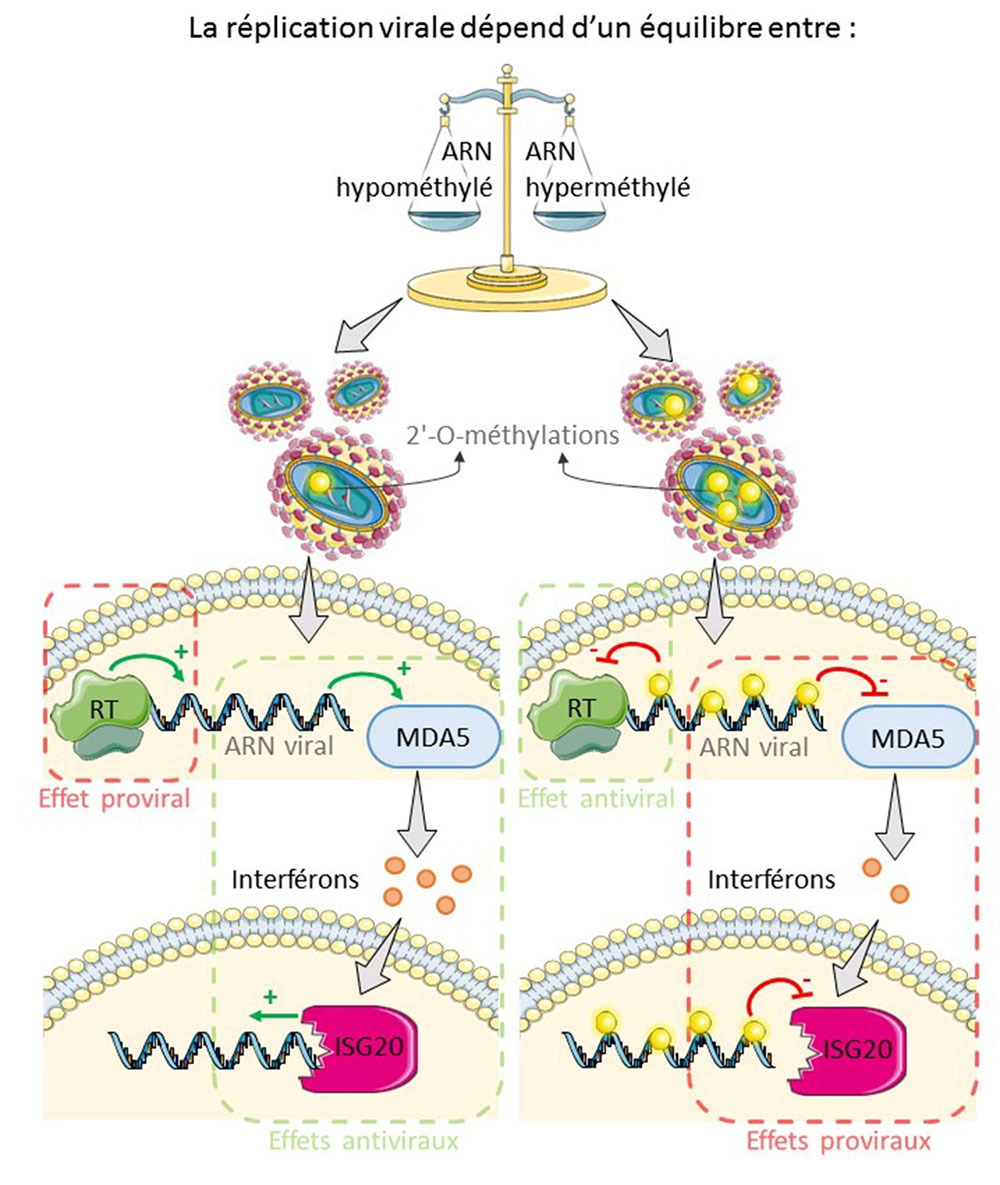
Figure : Représentation schématique des rôles proviraux et antiviraux liés aux modification épitranscriptomiques (2′-O-méthylations) de l’ARN du VIH-1. En absence de 2′-O-méthylation, la polymérase virale réalise plus efficacement la transcription inverse, cependant l’ARN viral est détecté par MDA5 qui stimule la production d’interférons, et l’exonucléase ISG20 induite par l’interféron dégrade le génome ARN viral. L’infection productive des cellules semble donc reposer sur un contrôle de la balance entre hypo- et hyperméthylation du génome ARN viral, induisant un équilibre entre les effet pro- et antiviraux des 2′-O-méthylations.
En savoir plus :
Alice Decombe, Olve Peersen, Priscila Sutto-Ortiz, Célia Chamontin, Géraldine Piorkowski, Bruno Canard, Sébastien Nisole, Etienne Decroly, Internal RNA 2′-O-methylation on the HIV-1 genome impairs reverse transcription, Nucleic Acids Research, 2023;, gkad1134, https://doi.org/10.1093/nar/gkad1134
Contact
Laboratoire
Architecture et fonction des macromolécules biologiques - AFMB (CNRS/Aix Marseille Université)
163 Avenue de Luminy
13009 Marseille