La cryo-microscopie électronique pour comprendre l’assemblage des ribosomes
Les ribosomes sont les machines moléculaires qui, dans toutes les cellules vivantes, décodent l’information génétique et la traduisent en protéines. Pour comprendre comment ces ribosomes s’assemblent, une équipe internationale de chercheurs a recouru à différentes approches intégrant génétique, analyses fonctionnelles et cryo-microscopie électronique. Cette dernière technique leur a permis de déterminer la structure atomique de précurseurs des ribosomes bloqués à des stades tardifs de leur maturation. Ils ont ainsi pu montrer comment des événements se produisant sur des sites distants des particules pré-ribosomiques sont interconnectés et synchronisés, pour permettre la synthèse de ribosomes fonctionnels. Cette étude est publiée dans la revue Nature Communications.
Les ribosomes sont d’énormes complexes moléculaires, subdivisés en une petite ("40S") et une grande ("60S") sous-unité, toutes deux composées d’ARN et de protéines. Présents dans les cellules de tous les organismes vivants, les ribosomes jouent le double rôle de maître d’œuvre et de chaîne d’assemblage, puisque la petite sous-unité lit l’information contenue par les ARN messagers et la transmet à la grande sous-unité, qui assure la synthèse des protéines correspondant à ce code génétique. Fabriquer des sous-unités ribosomiques parfaitement fonctionnelles est donc un enjeu majeur pour tous les organismes vivants.
Chez les eucaryotes, ce processus est très complexe : il s’effectue en de nombreuses étapes, qui ont lieu dans plusieurs compartiments successifs de la cellule. Il requiert également plus de 200 facteurs de maturation (protéines et complexes ribo-nucléoprotéiques) qui vont notamment permettre l’assemblage et le repliement corrects des ARN et protéines ribosomiques, mais dont le rôle exact reste à déterminer. Chez l’Homme, des défauts d’assemblage des sous-unités ribosomiques sont responsables de diverses maladies génétiques, comme des formes de cancers héréditaires ou d’anémies sévères. Il est donc capital de décrypter précisément toutes les étapes de l’assemblage des sous-unités ribosomiques, en conditions normales ou pathologiques.
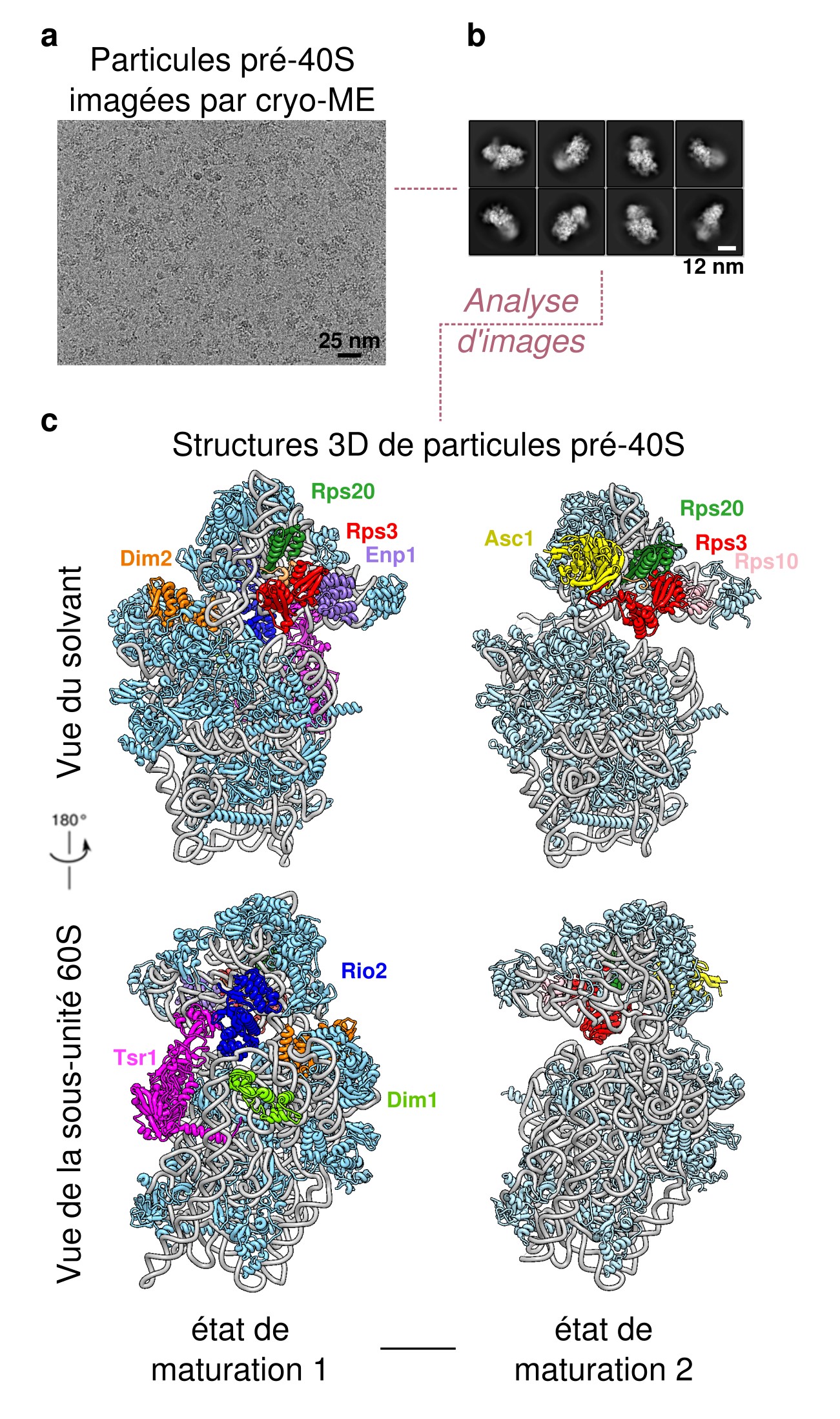
Pour mieux comprendre les dernières étapes de maturation de la petite sous-unité ribosomique, les scientifiques du Centre de Biologie Intégrative de Toulouse, en collaboration avec des équipes des universités de Regensburg (Allemagne), Fribourg (Suisse), et Graz (Autriche) ont utilisé la levure comme organisme modèle et introduit une mutation dans la protéine ribosomique Rps20, afin de bloquer les particules pré-40S à un stade tardif de leur maturation. Ils ont ensuite purifié ces particules, puis utilisé la cryo-microscopie électronique et l’analyse d’images pour déterminer leur structure 3D à une résolution quasi-atomique. Leurs résultats montrent que la mutation de Rps20 bloque la maturation de la particule à deux stades successifs. Couplée à des analyses génétiques et biochimiques, cette étude a également révélé le rôle de capteur/effecteur de Rps20 dans le processus d’assemblage de la petite sous-unité ribosomique. Ainsi, par sa position et sa conformation, Rps20 surveille des événements de maturation physiquement éloignés, et ne permet la progression vers les étapes finales de la maturation de la petite sous-unité ribosomique que si toutes les étapes antérieures, se produisant de part et d’autre de la structure de la particule pré-40S, ont été correctement effectuées.
Cette étude est une avancée importante dans la compréhension de l’assemblage des ribosomes, car peu de structures atomiques de particules pré-ribosomiques ont été résolues à ce jour. En outre, ces travaux, alliant l’analyse structurale par cryo-MET à un corpus d’analyses génétiques, moléculaires et biochimiques permettent de proposer un modèle global et dynamique des dernières étapes de maturation de la petite sous-unité ribosomique.
© C.Plisson-Chastang & B. Pertschy
Pour en savoir plus :
Conformational proofreading of distant 40S ribosomal subunit maturation events by a long-range communication mechanism.
Mitterer V, Shayan R, Ferreira-Cerca S, Murat G, Enne T, Rinaldi D, Weigl S, Omanic H, Gleizes PE, Kressler D, Plisson-Chastang C, Pertschy B.
Nat Commun. 2019 Jun 21;10(1):2754. doi: 10.1038/s41467-019-10678-z.