La composition nucléotidique des introns contribue directement à l'efficacité de leur épissage
L'ADN est enroulé autour de protéines pour former les nucléosomes. Les gènes y sont transcrits en ARN pré-messagers, composés d'exons et d'introns, ces derniers étant "épissés" pour ne conserver que les exons dans l'ARN messager final. Les nucléosomes sont préférentiellement positionnés au niveau des exons par rapport aux introns. Cette étude publiée dans la revue Genome Research montre que la composition nucléotidique des introns, et non pas le positionnement des nucléosomes, contribue directement à l’efficacité d’épissage des introns chez la paramécie.
Chez les eucaryotes, les nucléosomes ne sont pas situés au hasard le long du génome mais positionnés par rapport à la séquence d'ADN. Le positionnement des nucléosomes est très dynamique et sa régulation est cruciale pour contrôler l'accessibilité de la chromatine et un certain nombre de processus biologiques importants tels que la transcription et la maturation des ARN messagers (ARNm). Les nucléosomes sont préférentiellement positionnés dans les exons par rapport aux introns. Un nucléosome bien positionné pourrait ralentir l'ARN polymérase II et favoriser l'inclusion d'exon et l'épissage alternatif, ce qui suggère un rôle fonctionnel du positionnement des nucléosomes pendant la maturation de l'ARNm. Certaines études ont suggéré que la richesse en nucléotides GC des exons, et non le positionnement des nucléosomes en soi, est importante pour l'épissage des introns. A ce jour, aucune évaluation solide de la contribution du positionnement des nucléosomes à l'efficacité de l'épissage des introns n'a encore été rapportée.
Pour répondre à cette question, les chercheurs ont étudié l'effet du positionnement des nucléosomes sur l'efficacité de l'épissage en utilisant l’eucaryote unicellulaire cilié Paramecium tetraurelia comme modèle. Chez cet organisme, le macronoyau somatique est responsable de l’expression des gènes. Les 90 000 introns annotés dans le génome sont parmi les plus petits reportés chez les eucaryotes (entre 18 et 33 nucléotides de long, 25 nucléotides en moyenne). Les chercheurs ont déterminé la première carte d'occupation des nucléosomes du génome somatique de P. tetraurelia riche en nucléotides AT (>75%) et trouvé la plus petite longueur de répétition de nucléosomes (~151 bp) chez les eucaryotes. Leur étude révèle que les introns sont fréquemment associés à l'ADN inter-nucléosomal (riche en AT) et que les introns situés au bord des nucléosomes présentent une efficacité d'épissage plus élevée que ceux situés au centre. Les chercheurs ont ensuite démontré que la composition en nucléotides de l'intron, la teneur en GC en particulier, plutôt que le positionnement des nucléosomes, contribue directement à l'efficacité de l'épissage : un contenu élevé en nucléotides GC des introns est corrélé à une occupation en nucléosomes élevée et est associée à une faible efficacité d’épissage.
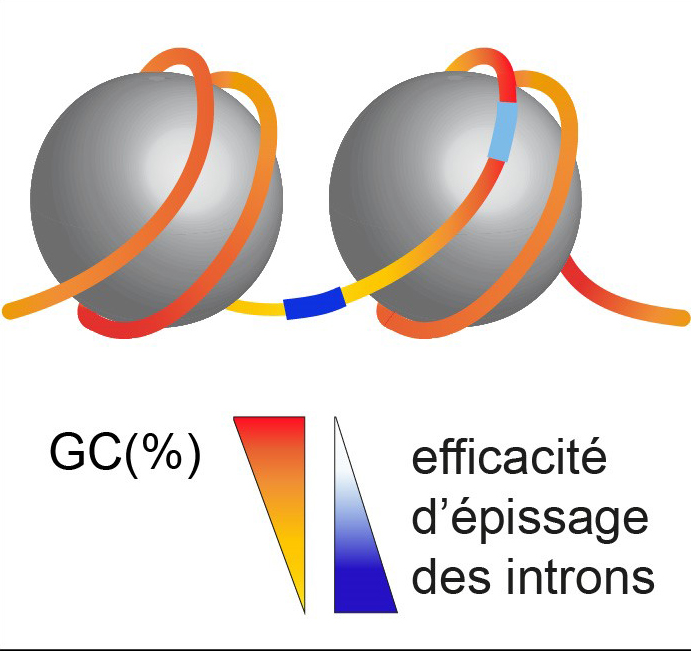
Figure : Chez l’eucaryote unicellulaire cilié Paramecium tetraurelia, un contenu élevé en nucléotides GC des introns est corrélé à une occupation en nucléosomes élevée et est associée à une faible efficacité d’épissage.
Pour en savoir plus :
GC content but not nucleosome positioning directly contributes to intron-splicing efficiency in Paramecium.
Gnan S, Matelot M, Weiman M, Arnaiz O, Guerin F, Sperling L, Betermier M, Thermes C, Chen CL, Duharcourt S.
Genome Research, 9 mars 2022. doi: 10.1101/gr.276125.121.
Contact
Laboratoires
Institut Jacques Monod (Université Paris Cité/CNRS)
15 rue Hélène Brion
75013 Paris
Institut Curie (CNRS/Université PSL/Sorbonne Université)
26 rue d’Ulm
75005 Paris
