Histoire évolutive de l’ultrarésistance aux antibiotiques de Mycobacterium tuberculosis
A l’origine de 1.5 millions de décès chaque année, la tuberculose est une cause première de mortalité due à une maladie infectieuse, seulement devancée par la COVID en 2020-2021. Une étude basée sur le séquençage génomique a déterminé les facteurs bactériens et certains évènements historiques impliqués dans l’émergence et la propagation internationale de souches multirésistantes aux traitements. Ces résultats, publiés dans la revue Nature Communications, soulignent la nécessité de déployer de nouveaux moyens de détection de résistance étendue.
La tuberculose multi-/ultra-résistante aux antibiotiques, contractée le plus souvent par transmission interhumaine, est la principale cause de mortalité humaine due à la résistance aux antimicrobiens. Des branches particulières d’une lignée de souches de Mycobacterium tuberculosis sont responsables de fortes prévalences de multirésistance en Eurasie, et présentent des distributions transnationales exceptionnelles.
Des scientifiques français et allemands ont déterminé les facteurs sous-jacents à l'émergence et à la propagation épidémique de la branche appelée W148 en analysant les génomes de plus de 700 souches de 23 pays. Les résultats permettent de dater un ancêtre commun déjà résistant à l’isoniazide et la streptomycine au début des années 60, concordant avec l'introduction de ces antibiotiques à cette époque. Des expansions épidémiques successives ont suivi à la fin des années 1980 et à la fin des années 1990, coïncidant avec des changements socio-économiques majeurs de l'ère post-soviétique. Ces souches ont évolué vers des niveaux de (pré-)ultrarésistance en moins de deux décennies, accumulant des mutations de résistance à jusqu'à 11 médicaments. La transmission des souches les plus résistantes est associée à l'acquisition de mutations compensant le coût métabolique dû aux mutations de résistance. Toutes les souches W148 possèdent également une mutation d’hypervirulence, et certaines d’entre elles ont acquis des mutations additionnelles pouvant encore favoriser le développement de résistances supplémentaires.
Cet arsenal génétique exceptionnel et la large distribution géographique de ces souches constituent une «tempête parfaite», mettant en péril le succès de l'introduction des nouveaux traitements de tuberculose multirésistante. Le déploiement à plus grande échelle de nouveaux tests à spectre diagnostique étendu, tels le test Deeplex Myc-TB récemment développé par GenoScreen avec le concours d’un chercheur du CNRS, sera essentiel pour combattre la propagation de telles souches hautement résistantes.
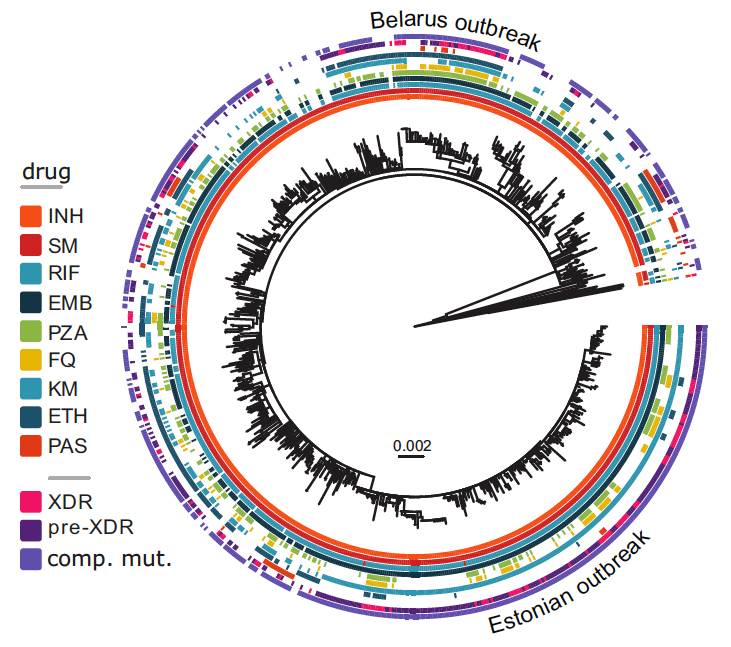
Figure : Arbre phylogénétique basé sur le séquençage des génomes de 720 souches W148 et 12 souches externes apparentées. Les différentes couronnes colorées montrent, de l’intérieur vers l’extérieur, la présence (case pleine) ou l’absence (case vide) de mutations conférant une résistance à différents médicaments (INH isoniazide, SM streptomycine, RIF rifampicine, EMB éthambutol, PZA pyrazinamide, FQ fluoroquinolones, KM kanamycin, ETH éthionamide et PAS acide para-aminosalicylique), les profils d’antibiorésistance associés (XDR ultrarésistance et pre-XDR pré-ultrarésistance), et la présence de mutations compensatrices de coût métabolique dû aux mutations d’antibiorésistance. Des groupes de variants épidémiques particulièrement localisés en Biélorussie et en Estonie sont signalés.
Pour en savoir plus :
Transcontinental spread and evolution of Mycobacterium tuberculosis W148 European/Russian clade toward extensively drug resistant tuberculosis.
Merker M, Rasigade JP, Barbier M, Cox H, Feuerriegel S, Kohl TA, Shitikov E, Klaos K, Gaudin C, Antoine R, Diel R, Borrell S, Gagneux S, Nikolayevskyy V, Andres S, Crudu V, Supply P, Niemann S, Wirth T.
Nature Communications 30 aout 2022 doi: 10.1038/s41467-022-32455-1
Contact
Laboratoire
Centre d’infection et d’immunité de Lille - CIIL (Université de Lille/CNRS/Inserm/CHU Lille/Institut Pasteur de Lille)
1, rue du Professeur Calmette
59019 Lille, France