Exploiter le « code chimique » de l’ARN pour établir une carte d’identité tumorale
L’utilisation de biomarqueurs est fondamentale pour établir avec précision la nature et le degré d'agressivité d'un cancer, ou encore pour adapter le traitement en fonction des caractéristiques moléculaires. Les altérations épigénétiques (qui modulent l’activité du gène mais pas sa séquence d’ADN) sont utilisées comme indicateurs cliniques à cause de leur valeur prédictive ou pronostique. Dans un article publié dans la revue Analytical Chemistry, les scientifiques présentent une méthode nouvelle, couplant l’analyse des modifications épigénétiques de l’ARN à de l’intelligence artificielle, afin de déterminer le stade d’un cancer du cerveau (le gliome) avec une efficacité inégalée.
Les ARN sont les molécules intermédiaires porteuses de l’information génique. Ils sont essentiellement composés de l’agencement de quatre bases nucléiques (adénine, uracile, cytosine, et guanine) mais peuvent être également « décoré » de marques chimiques modifiant ces bases. Regroupées autour du terme « épitranscriptome », ces modifications chimiques représentent un niveau supplémentaire de contrôle de l’expression génique. Plus de 150 marques chimiques ont été découvertes dans divers organismes et espèces d’ARN, formant un code complexe, dynamique et représentatif d’un état cellulaire ou tissulaire. Un nombre croissant de ces marques chimiques est associé à des pathologies humaines et près de la moitié des enzymes qui contrôlent ces voies présentent des altérations fonctionnelles dans des maladies neurologiques, développementales, métaboliques, cardiovasculaires et les cancers. Dans un contexte tumoral, ces modifications peuvent favoriser l’évolution de la maladie, sa dissémination dans l’organisme, l’acquisition de résistance aux thérapies conventionnelles, voire la récidive.
Dans une approche pluridisciplinaire (biologie, chimie, médecine, informatique) les scientifiques ont conçu une méthode combinant l'analyse multiplexe de l’épitranscriptome par spectrométrie de masse avec une analyse statistique multivariée et des approches d'apprentissage automatique (IA). Leur objectif était de mettre en évidence une combinaison de marques, c’est à dire une « signature », permettant de discriminer des stades tumoraux, de guider la médecine de précision et d’améliorer le diagnostic de la maladie.
Cette approche a été appliquée pour analyser le tissu tumoral d’une cohorte de patients adultes atteints de gliome diffus (de bas et hauts stades). En effet, les stades 2 (bas stade) et 3 (haut stade) sont difficiles à discriminer par les méthodes classiques utilisées en routine clinique. De ce fait, le diagnostic de la maladie peut nécessiter plusieurs semaines. Cette étude révèle l'existence d'une « signature épitranscriptomique » permettant de discriminer et de prédire les stades de gliome, tout particulièrement les stades 2 et 3, en quelques heures et avec une précision inégalée. Cette étude, qui fait l’objet d’un brevet, établit la preuve de concept que le paysage épitranscriptomique évolue au cours de la progression tumorale, très certainement afin de permettre aux cellules cancéreuses de s’adapter à un environnement hostile à leur développement. De ce fait, elle ouvre de nouvelles perspectives dans le domaine du profilage moléculaire des tumeurs et la médecine de précision.
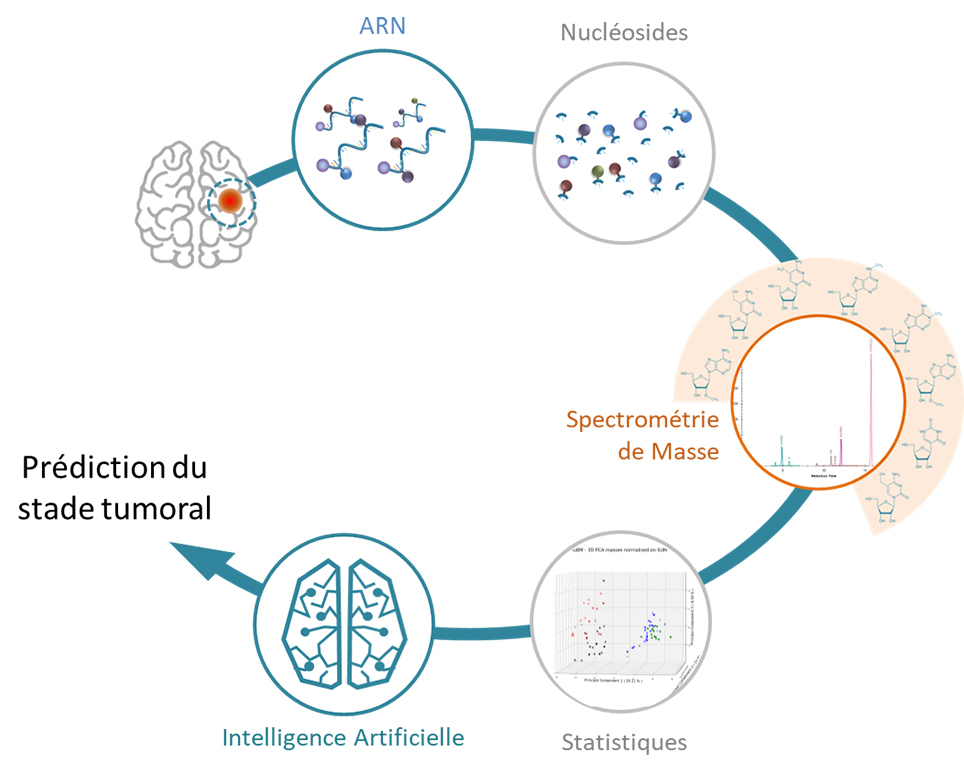
Figure : Vue globale de la méthode de profilage épitranscriptomique. L’ARN total est extrait de l’échantillon tumoral, puis dégradé en nucléosides. Ceux-ci sont analysés par spectrométrie de masse, permettant d’identifier et de quantifier ceux comportant des marques chimiques. Les données sont ensuite soumises à une analyse statistique. L’exploitation de ces données par l’intelligence artificielle permet de prédire le stade tumoral du tissu cancéreux.
Pour en savoir plus :
Multivariate Analysis of RNA Chemistry Marks Uncovers Epitranscriptomics-Based Biomarker Signature for Adult Diffuse Glioma Diagnostics.
S. Relier, A. Amalric, A. Attina, I.B. Koumare, V. Rigau, F. Burel Vandenbos, D. Fontaine, M. Baroncini, J.P. Hugnot, H. Duffau, L. Bauchet, C. Hirtz, E. Rivals, and A. David.
Analytical Chemistry 6 septembre 2022. doi: 10.1021/acs.analchem.2c01526.
Contact
Laboratoires
Institut de génomique fonctionnelle - IGF (CNRS/Inserm/Université de Montpellier)
141 Rue de la Cardonille
4094 Montpellier
Laboratoire d'informatique, de robotique et de microélectronique de Montpellier - LIRMM (CNRS/Université de Montpellier)
161 Rue Ada
4095 Montpellier
Institute for regenerative medicine & biotherapy - IRMB (Inserm/Université de Montpellier)
80 avenue Augustin Fliche
34295 Montpellier