Evolution et fonction de ciseaux moléculaires : les topoisomérases et la méiose
Lors de la méiose, de nombreux échanges entre chromosomes maternels et paternels se produisent. Ces événements de recombinaison procèdent par un mécanisme de cassure et de réparation de l’ADN lors de la prophase de la première division de méiose. Ces cassures sont produites par des enzymes apparentées aux topoisomérases. Dans deux études publiées dans Molecular Biology and Evolution et Nature communications, les scientifiques ont contribué à comprendre comment cette activité s’est spécialisée au cours de l’évolution pour se retrouver impliquée dans la reproduction sexuée en analysant l’origine évolutive de ces protéines.
La majorité des eucaryotes se reproduit par reproduction sexuée, et met en œuvre un mode de division spécialisé, la méiose, pour générer des cellules haploïdes. En prophase de première division de méiose, de nombreux événements de recombinaison procèdent par un mécanisme de formation et réparation de cassures double-brins de l’ADN (CDBs). Cette étape est un fort risque d’instabilité du génome. Pour être fidèlement réparées, les CDBs doivent être régulées en fréquence, localisation et cinétique par des mécanismes encore mal compris. Ces CDBs sont formées par une activité de type topoisomérase de type II par les protéines SPO11 et TOPOVIBL, qui sont des orthologues des sous-unités TopoVIA et TopoVIB respectivement, de la topoisomérase TopoVI, déjà présente chez les archées. Comment ces orthologues ont-ils évolué et comment leur activité biochimique s’est-elle modifiée pour adopter une fonction de cassure de l’ADN en méiose reste énigmatique. Une grande conservation évolutive de la protéine SPO11 a été identifiée chez la plupart des eucaryotes. Dans un premier article, les scientifiques ont analysé la phylogénie de TOPOVIBL et mis en évidence une grande diversité de cette protéine, en particulier au niveau du domaine qui, chez TopoVIB, est impliqué dans la fixation et l’hydrolyse de l’ATP. Un motif conservé chez les métazoaires dans la région carboxy-terminale de TOPOVIBL a également été identifié et interagit avec la protéine REC114 de souris, connue pour être indispensable pour la formation des CDBs en méiose.
Dans un deuxième article, les scientifiques ont donc testé le rôle fonctionnel de cette interaction, en introduisant des mutations ponctuelles dans le gène de souris codant pour TOPOVIBL qui diminuent ou abolissent l’interaction avec REC114. Ces mutations ont pour conséquence une diminution de l’activité globale des CDBs dans l’ovocyte mais uniquement dans les régions sous-télomériques dans le spermatocyte, avec un retard de formation dans l’ensemble du génome. L’ensemble de ces données permettent d’établir un modèle structural de SPO11/TOPOVIBL en association avec REC114 qui pourrait stabiliser l’hétérotétramère prédit par le modèle. REC114 apparait ainsi un régulateur de SPO11/TOPOVIBL dont l’activité pourrait être en outre modulée par les propriétés épigénétiques spécifiques de l’ovocyte et du spermatocyte.
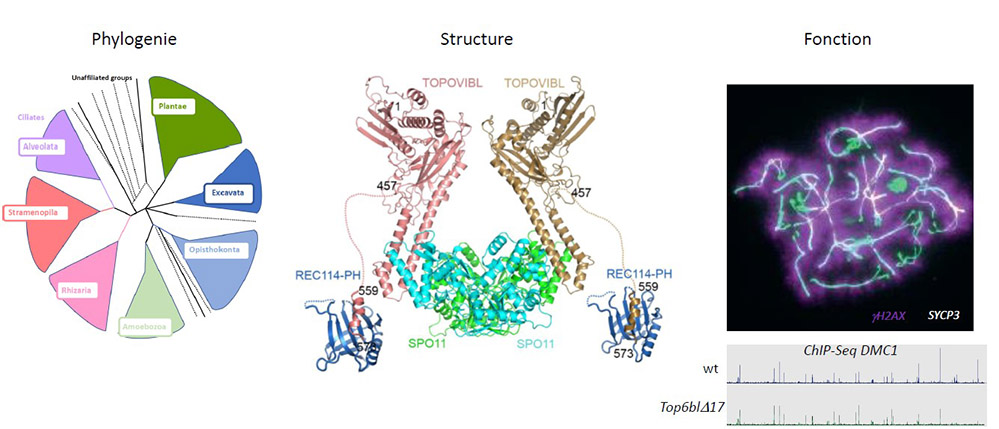
Figure : A gauche : La majorité des eucaryotes se reproduit par reproduction sexuée. Alors que le gène Spo11 est présent et conservé chez la plupart des eucaryotes, le gène codant pour TOPOVIBL est extrêmement divergent, voire absent dans certains taxa.
Au centre : Des données de structures et de modélisation proposent que SPO11 forme un hétérotétramère avec TOPOVIBL, qui interagit avec REC114.
A droite : Des analyses cytologiques et génomiques sur des spermatocytes et ovocytes de souris mettent en évidence le rôle de l’interaction entre TOPOVIBL et REC114.
Pour en savoir plus :
Evolution and Diversity of the TopoVI and TopoVI-like Subunits With Extensive Divergence of the TOPOVIBL subunit.
Brinkmeier, J., Coelho, S., de Massy, B., and Bourbon, H.M.
Molecular Biology and Evolution, 2022. DOI: 10.1093/molbev/msac227
TOPOVIBL-REC114 interaction regulates meiotic DNA double-strand breaks.
Nore, A., Juarez-Martinez, A.B., Clement, J., Brun, C., Diagouraga, B., Laroussi, H., Grey, C., Bourbon, H.M., Kadlec, J., Robert, T., and de Massy, B.
Nature Communications, 2022. DOI:https://doi.org/10.1038/s41467-022-34799-0
Contact
Laboratoire
Institut de génétique humaine – IGH (CNRS/Université de Montpellier)
141 rue de la cardonille
34396 Montpellier cedex 05
