Découverte d'un nouveau composant dans la construction de l’enveloppe des entérobactéries, un potentiel renforcé pour la découverte de nouveaux antibiotiques
Dans un article publié dans la revue Nature communications, les scientifiques ont identifié un nouveau composant impliqué dans la formation de l’enveloppe des entérobactéries. Cette barrière de protection rend la lutte contre les espèces pathogènes difficile. L'étude révèle ainsi un potentiel « talon d’Achille » important pour la découverte de nouveaux antibiotiques.
Les entérobactéries sont des bacilles Gram négatifs présents essentiellement dans la flore intestinale des mammifères. Elles comprennent les genres bactériens Escherichia, Salmonella, Klebsiella et Yersinia, parmi lesquels figurent des espèces qui représentent une des principales causes de mortalité par infection bactérienne. Cette menace est renforcée par l'acquisition de mécanismes de résistance aux thérapies antibiotiques traditionnelles, ce qui fait de la recherche de nouveaux antibiotiques une priorité essentielle pour les systèmes de santé publique dans le monde.
Identifier de nouvelles cibles moléculaires pour développer des thérapies antimicrobiennes
Les lipopolysaccharides sont des glycolipides exposés à la surface des bactéries Gram-négatives et fonctionnent comme des facteurs de virulence. Ils forment une barrière imperméable aux composés hydrophobes permettant aux bactéries de résister à de nombreux agents antimicrobiens et détergents. Les lipopolysaccharides peuvent également se comporter comme des endotoxines qui, une fois libérée par les bactéries, sont capables de stimuler le système immunitaire de l'hôte. Le transport des lipopolysaccharides vers la surface cellulaire est un processus essentiel chez la plupart des bactéries Gram-négatives. Ce processus n'a été caractérisé que récemment et recèle encore de nombreux secrets. Comprendre le processus de transport des lipopolysaccharides est particulièrement important dans le contexte de lutte contre les bactéries pathogènes. En effet, cela permettrait d'identifier de nouvelles cibles moléculaires pour développer des thérapies antimicrobiennes.
L'assemblage final des lipopolysaccharides à la surface des cellules bactériennes est assuré par un complexe de protéines appelé translocon, qui assure le transport de ces molécules à travers la membrane. Jusqu’à présent, seules les protéines LptD et LptE définissaient le translocon des lipopolysaccharides. Dans cette publication, les scientifiques ont découvert une lipoprotéine comme nouvelle pièce de cet outil dans une bactérie appelée Escherichia coli. Ils l'ont appelée LptM (pour LipoPolysaccharide Translocon Maturation)
LptM pour amorcer la translocation des lipopolysaccharides
Des techniques de génétique, de biochimie et de prédiction structurale ont permis de révéler la fonction de LptM. En effet, cette dernière interagit avec des sites du translocon connus pour lier les lipopolysaccharides. Cette interaction active le translocon et ouvre ainsi une porte moléculaire qui faciliterait le passage des lipopolysaccharides vers la surface cellulaire. Les scientifiques proposent un modèle dans lequel LptM, en imitant une molécule de lipopolysaccharides, amorce leur translocation. Cette lipoprotéine est largement présente chez les entérobactéries et d'autres bactéries à Gram négatif, ce qui rend ces résultats pertinents pour de nombreuses espèces bactériennes.
Cette étude élargit notre compréhension d'un processus essentiel dans la formation de l'enveloppe bactérienne, apportant un soutien important à la recherche de nouveaux agents antimicrobiens.
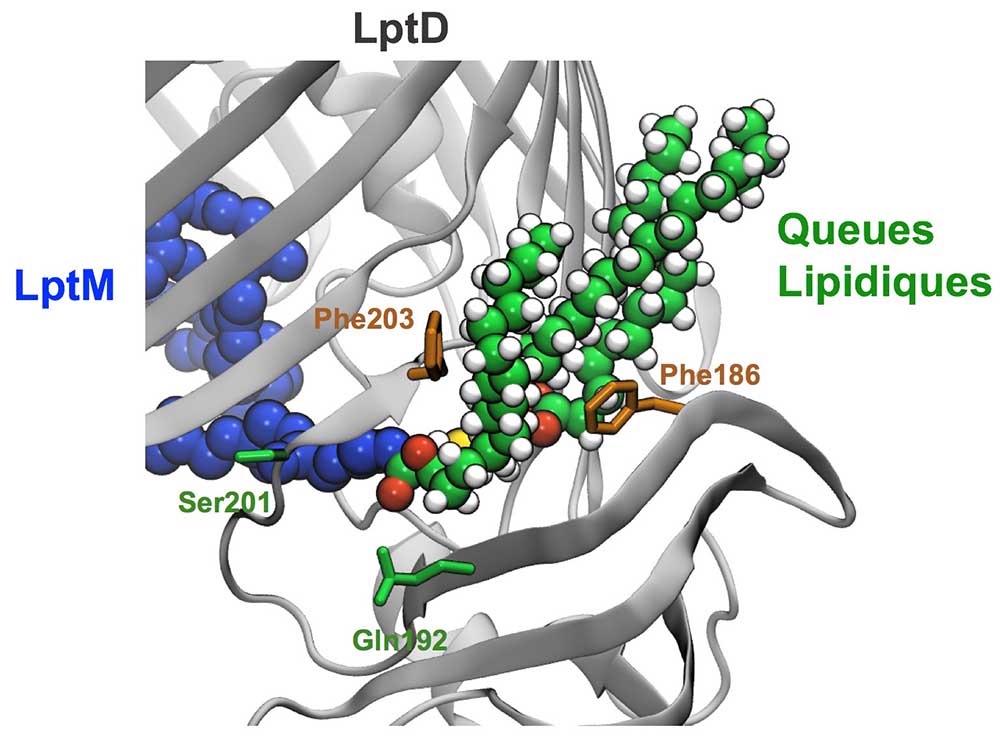
Figure : Vue rapprochée du site de transport des lipopolysaccharides dans la structure de LptD : Les essais biochimiques et la prédiction structurale montrent que la lipoproteine LptM occupe avec ses queues lipidiques le site de transport des lipopolysaccharides
En savoir plus :
LptM promotes oxidative maturation of the lipopolysaccharide translocon by substrate binding mimicry. Authors : Yiying Yang, Haoxiang Chen, Robin A. Corey, Violette Morales, Yves Quentin, Carine Froment, Anne Caumont-Sarcos, Cécile Albenne, Odile Burlet-Schiltz, David Ranava, Phillip J. Stansfeld, Julien Marcoux, Raffaele Ieva Nature Communications https://www.nature.com/articles/s41467-023-42007-w PMID: 37821449
Contact
Laboratoire
Laboratoire de microbiologie et génétique moléculaire - LMGM (CNRS/Université Toulouse Paul Sabatier)
Centre de biologie intégrative (CBI) de Toulouse
118 route de Narbonne
31062 Toulouse
