Dans l’intimité moléculaire du virus de l’hépatite D
L'hépatite delta ou hépatite D, la forme la plus sévère des hépatites virales, reste mal comprise à l’échelle moléculaire. Dans deux articles publiés dans Proceedings of the National Academy of Sciences (PNAS) et Journal of the American Chemical Society (JACS), des scientifiques ont étudié la structure de l’unique protéine virale et ont identifié les régions impliquées dans l'interaction avec le génome viral. Ces résultats ouvrent la voie à de nouvelles stratégies thérapeutiques ciblant l’assemblage du virus.
Un virus redoutable à l’arsenal minimal
Responsable de la forme la plus sévère d’hépatite virale chronique, le virus de l’hépatite D (VHD) infecte plus de 165 millions de personnes dans le monde. C’est un virus satellite du virus de l’hépatite B (VHB). En effet, l’infection ne se produit que si la personne est déjà infectée par le VHB. Le VHD est doté d’un génome extrêmement réduit, un petit ARN circulaire. Il ne code que pour une seule protéine virale, l’antigène delta (HDAg) présentant deux isoformes, L-HDAg et S-HDAg, essentiels à sa réplication. Si la structure de son extrémité N-terminale a été résolue auparavant, celle de la protéine entière restait jusque-là un mystère.
Une approche innovante de production pour étudier la structure complète de la protéine virale
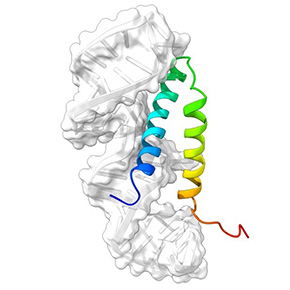
La protéine virale entière étant insoluble, elle ne peut pas être produite par les méthodes habituellement utilisées par les biochimistes, comme l’expression en bactérie. Les scientifiques ont donc eu recours à une production acellulaire, exploitant la machinerie de synthèse protéique des extraits de germes de blé. Cette méthode permet aujourd’hui d’obtenir suffisamment de protéines pour des analyses par spectroscopie RMN du solide à rotation rapide, afin d’étudier le domaine d’assemblage du VHD dans le contexte de la protéine entière. Dans une première étude parue dans JACS, cette approche a permis aux scientifiques d’examiner la structure du domaine d’assemblage (oligomérisation) du VHD dans le contexte de la protéine complète. Alors que ce domaine avait déjà été caractérisé isolément par cristallographie aux rayons X, les données de RMN du solide ont montré que sa structure est conservée au sein de la protéine entière et que la formation de dimères persiste. Plus encore, cette étude a révélé que le domaine d’assemblage est la seule région rigide de la protéine, tandis que le domaine C-terminal, soluble à l’état isolé, reste flexible dans le contexte de la protéine complète.
Caractérisation fine du domaine C-terminal et de ses interactions avec l’ARN par RMN en solution
Dans une seconde étude publiée dans la revue PNAS, c’est la RMN en solution qui est mobilisée pour analyser la partie C-terminale, notamment à travers une forme tronquée nommée SΔ60. Les analyses ont révélé que SΔ60 contient deux régions désordonnées, séparées par un motif structuré de type hélice-boucle-hélice (HLH), conservé au sein de la protéine complète. Ce motif, constitué de deux hélices antiparallèles stabilisées par des interactions électrostatiques et hydrophobes, contient des domaines riches en arginine fortement conservés chez les huit génotypes du VHD. Il comporte aussi un groupe d’acides aminés chargés négativement en surface, mimant potentiellement une zone reconnue par des facteurs de remodelage de la chromatine, ce qui pourrait favoriser la réplication du virus.
L’ARN viral complet du VHD a été transcrit in vitro, puis caractérisé par microscopie à force atomique (AFM) et microscopie électronique, afin d’étudier son interaction avec SΔ60. Les expériences de RMN en solution ont montré que les hélices du motif HLH constituent le site principal de liaison à l’ARN, tandis que les régions désordonnées, notamment en C-terminal, ne participent pas directement à l’interaction. La liaison est forte, avec une affinité submicromolaire, et implique environ 120 molécules de SΔ60 par ARN viral.
En combinant RMN du solide et RMN en solution, les scientifiques ont ainsi mis en lumière la dualité structurale de la protéine virale : un domaine d’assemblage rigide essentiel à la formation de multimères, et un domaine C-terminal flexible, dont une partie structurée joue un rôle clé dans l’interaction avec l’ARN et la réplication du virus.
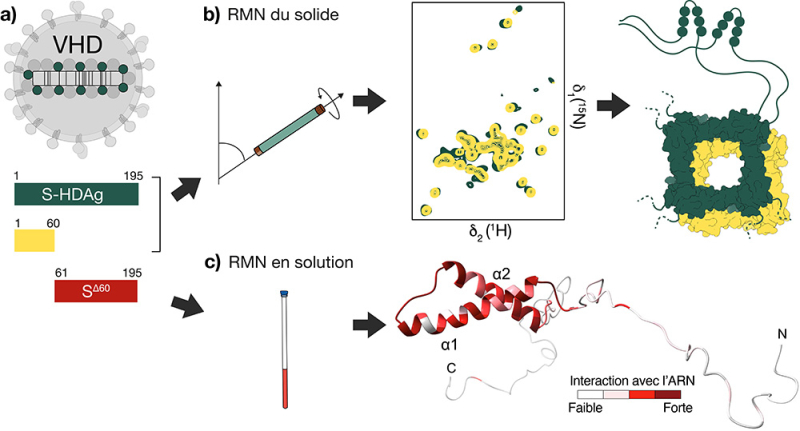
Figure : a) Schéma représentant le virus de l’hépatite D (VHD), et les différents domaines étudiés. b) La structure du domaine d’assemblage de la protéine HDAg a été déterminée par résonance magnétique nucléaire (RMN) du solide. c) La structure tridimensionnelle de SΔ60 a été déterminée par RMN en solution. Le modèle structurel illustre l’interaction entre le motif HLH de SΔ60 et une région de l’ARN viral.
En savoir plus : Y. Yang, L. Delcourte, C. van Belleghem, S. Fonte, K. Gerard, S. Baconnais, M. Callon, E. Le Cam, M. Fogeron, M. Levrero, C. Faivre-Moskalenko, A. Böckmann, & L. Lecoq, Structure and nucleic acid interactions of the SΔ60 domain of the hepatitis delta virus small antigen, Proc. Natl. Acad. Sci. U.S.A. 122 (19) e2411890122, https://doi.org/10.1073/pnas.2411890122 (2025).
En savoir plus : Yang, Y, Fogeron, M.-L., Malär, A.A., Lecoq, L., Barnes, A.B., Meier, B.H., Böckmann, A., and Callon, M. (2024) Hepatitis Delta Antigen Retains the Assembly Domain as the Only Rigid Entity. Journal of the American Chemical Society, 146 (43), 29531-29539 DOI: 10.1021/jacs.4c09409
Contact
Laboratoire
Microbiologie moléculaire et biochimie structurale - MMSB (CNRS/Université Claude Bernard)
7 passage du Vercors
69367 Lyon