Coopération cruciale pour la maturation des ARN de transfert chez les plantes
Les ARNt, ou ARN de transfert, sont des petits ARN universels essentiels à la traduction. Dans une étude publiée dans la revue Nature Plants, les scientifiques ont identifié un complexe fonctionnel singulier entre une enzyme de modification des ARNt et une nucléase dans le noyau de la plante modèle Arabidopsis. Ces deux enzymes coopèrent lors des premières étapes de la biogenèse des ARNt.
Les ARN de transfert sont des petits ARN non codants omniprésents qui assurent la jonction entre l’information génétique contenue dans les ARNm, ou ARN messagers, et les séquences protéiques lors de la traduction. Ces molécules adaptatrices, chacune chargée d'un acide aminé spécifique, se lient à l'ARNm au niveau de la petite sous-unité du ribosome, jouant ainsi un rôle essentiel dans le processus de traduction. Comme tous les ARN, les ARNt sont produits sous forme de précurseurs. L’une des étapes cruciales de leur maturation implique l’élimination de séquences additionnelles en 5’. Cette activité est catalysée par une famille d'endoribonucléases connue sous le nom de RNase P. Chez les plantes, l’activité RNase P est exclusivement assurée par les protéines « PRORP ». Bien que l'évolution et le mode d'action des protéines PRORP aient été largement étudiés, l’intégration de leurs fonctions à d'autres processus cellulaires reste peu connue.
Dans un article publié récemment dans la revue Nature Plants, les scientifiques ont élucidé le réseau d’interactions de la protéine PRORP2, localisée dans le noyau de la plante modèle Arabidopsis. Par une approche d’immunoprécipitation, ils ont identifié les méthyltransférases TRM1A et TRM1B comme étant les principaux partenaires protéiques de PRORP2. Ces protéines, également localisées dans le noyau, sont des enzymes de modification de l’ARN qui catalysent la formation de N2,N2-diméthylguanosine (m22G) sur le résidu 26 des ARNt cytosoliques en cours de maturation. Grâce à une approche « tRNAseq » à l'échelle du transcriptome, les scientifiques ont de plus montré que TRM1A et B sont responsables de la modification m22G26 de 70 % des ARNt cytosoliques in vivo et que les mutants TRM1A/B présentent une altération de l'activité RNase P. Dans l’ensemble, ces résultats apportent un éclairage nouveau sur les premières étapes de la biogenèse des ARNt chez les plantes, mettant en évidence l’importance de la coopération fonctionnelle entre TRM1A/B et la RNase P.
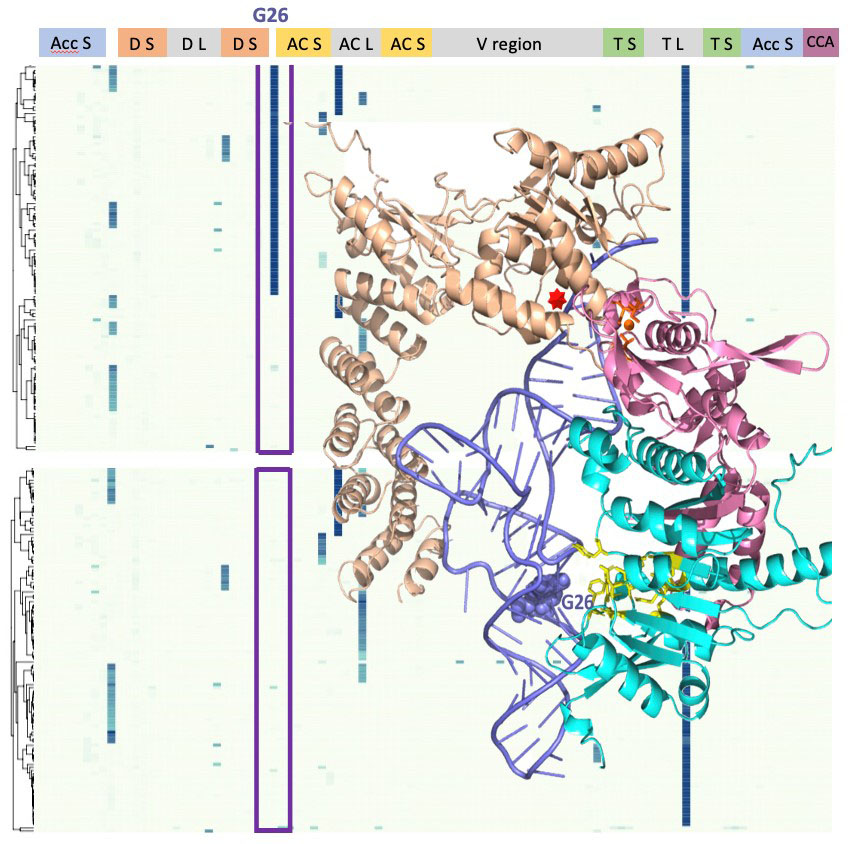
Figure : Modèle du complexe formé par PRORP (brun), un pré-ARNt (en bleu) et TRM1A (cyan et rose), surimposé sur une heatmap représentant la fréquence de mésappariements entre les résultats de tRNAseq et les séquences génomiques dans des échantillons de plante sauvage (en haut) et de double mutant TRM1A/B (en bas). Ce graphique permet de visualiser l’absence de modification en position G26 dans le mutant pour l’ensemble des ARNt cytosoliques.
En savoir plus :
Arrivé, M., Bruggeman, M., Skaltsogiannis, V. et al. A tRNA-modifying enzyme facilitates RNase P activity in Arabidopsis nuclei. Nat. Plants (2023). https://doi.org/10.1038/s41477-023-01564-0
Contact
Laboratoire
Institut de biologie moléculaire des plantes - IBMP (CNRS)
12, rue du général Zimmer,
67084 Strasbourg, France