Contrôle qualité de l'assemblage des ribosomes bactériens par le dégradosome d’ARN
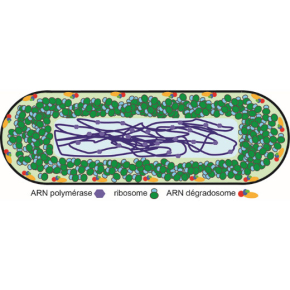
Les dégradosomes d’ARN sont des complexes protéiques qui, en traitant et dégradant les ARNs, façonnent le transcriptome pour assurer le contrôle de l’expression des gènes. Chez beaucoup de bactéries Gram-négatives les dégradosomes d’ARN sont des protéines localisées au niveau de la membrane interne de la bactérie. La raison de cette localisation n’était pas comprise.
En utilisant des mutants qui possèdent des dégradosomes libres dans le cytoplasme, les scientifiques, dans un article publié dans la revue Plos Biology, démontrent que cette localisation est importante pour préserver la qualité de l’assemblage des ribosomes.
La maturation et la dégradation des ARN façonnent le transcriptome en générant des molécules stables nécessaires à la traduction (ARNr et ARNt) et en facilitant le turnover des ARNm nécessaire au contrôle post-transcriptionnel de l'expression des gènes. Chez les bactéries et dans le chloroplaste des plantes, les dégradosomes d’ARN sont des complexes multi-enzymes qui maturent et dégradent les ARN. Les dégradosomes d’ARN contenant la RNase E sont attachés à la membrane cytoplasmique interne chez une large famille de bactéries Gram-négatives (β- et γ-protéobactéries). Jusqu'à présent, la raison de leur localisation à la membrane n'était pas comprise.
Dans cette étude, les scientifiques montrent qu'une souche mutante d'Escherichia coli, dans laquelle le dégradosome d’ARN est localisé à l'intérieur de la cellule, accumule des particules 20S et 40S qui sont des précurseurs de l'assemblage de ribosomes défectueux. Leur composition protéique est anormale et ils contiennent des précurseurs des ARNr clivés par la RNase E. Ils en ont conclu que les dégradosomes d’ARN situés à l'intérieur de la cellule dans la souche mutante interfèrent avec l’assemblage des sous-unités ribosomales
Pour mieux comprendre le mécanisme, ils ont montré in vitro, que les ARNr contenus dans les ribosomes intacts résistent au clivage par la RNase E, alors que les ARNr sans protéines sont facilement dégradés. Ceci démontre que la structure compacte des ribosomes matures protège les ARNr de la dégradation par la RNase E.
La plupart des opérons des ARNr sont à proximité dans le nucléoïde, suggérant l'existence d'un nucléole bactérien. Les premières étapes de l'assemblage des ribosomes bactériens impliquent le repliement co-transcriptionnel des ARNr et la liaison des protéines ribosomales (r-protéines) dans le nucléoïde. De multiples voies d'assemblage agissent en parallèle dans des blocs coopératifs de repliement des ARNr pour constituer les domaines structurels des sous-unités ribosomales matures. Étant donné la complexité du processus d'assemblage des ribosomes, il n'est pas surprenant que des intermédiaires défectueux apparaissent occasionnellement.
Les dégradosomes d’ARN fixés à la membrane dans les cellules de type sauvage contrôleraient la qualité de l'assemblage des ribosomes après la libération des intermédiaires du nucléoïde. L'attachement du dégradosome d’ARN à la membrane cytoplasmique interne des bactéries empêche la dégradation inutile des précurseurs d'ARNr, expliquant ainsi pourquoi cet attachement est conservé dans toutes les β- et γ-protéobactéries. L’identification des molécules qui interfèrent avec la fixation du dégradosome d’ARN à la membrane cytoplasmique interne pourrait avoir des applications en pharmacologie et en biotechnologie.
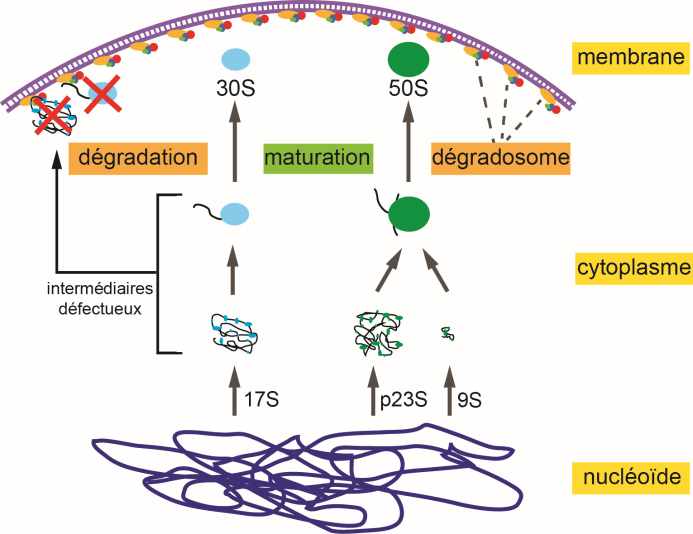
Figure : Les dégradosomes d’ARN fixés à la membrane cytoplasmique interne contrôlent la qualité de l'assemblage des ribosomes après la libération des intermédiaires du nucléoïde.
Pour en savoir plus :
Attachment of the RNA degradosome to the bacterial inner cytoplasmic membrane prevents wasteful degradation of rRNA in ribosome assembly intermediates.
Hadjeras L, Bouvier M, Canal I, Poljak L, Morin-Ogier Q, Froment C, et al.
PLoS Biology. 5 janvier 2023. DOI:https://doi.org/10.1371/journal.pbio.3001942.
Contact
Laboratoire
Toulouse biotechnology institute - TBI (Université de Toulouse/CNRS)
135 rue de Rangueil
31077 Toulouse