Comment les variants de SARS-CoV-2 sont-ils sélectionnés ?
Les variants préoccupants de SARS-CoV-2 comme Alpha, Delta ou Omicron se sont répandus rapidement dans le monde entier. Leur succès évolutif dépend de façon complexe du nombre de personnes qu’un individu atteint par le virus infecte à son tour, du timing de ces infections, et de la fréquence des contacts. Un article paru dans la revue eLife clarifie ces relations grâce à la modélisation mathématique, et permet de mieux comprendre les caractéristiques de ces variants.
Les mutations du virus SARS-CoV-2 ont conduit à l’émergence de variants préoccupants, comme Alpha, Delta et plus récemment Omicron. Ces souches portent des modifications génétiques qui permettent au virus de se propager plus facilement et peuvent modifier la gravité de la maladie qu'il provoque. Ces variants ont entraîné des augmentations du nombre d’infections, appelées vagues épidémiques, dans de nombreuses régions du monde.
Le suivi des nouveaux variants préoccupants est crucial pour anticiper ces dynamiques épidémiques. Pour ce faire, les scientifiques estiment le nombre d’individus qu’une personne atteinte du virus peut infecter, également connu sous le nom de « nombre d’infection secondaire ». Ils peuvent également estimer le timing où un individu infecté transmet le virus à d’autres au cours de l’infection. Ces deux mesures aident à déterminer la vitesse et l’ampleur d’une épidémie causée par un nouveau variant. Plus le nouveau variant infecte de personnes et plus il se propage rapidement, plus il est susceptible de remplacer les souches existantes du virus.
Jusqu’à présent, la plupart des études ont supposé que le meilleur taux de croissance d’un nouveau variant dépend uniquement du plus grand nombre d’infections secondaires qu’il engendre. Le timing des infections secondaires n’est souvent pas pris en compte. Pour y remédier, les scientifiques ont construit un modèle mathématique qui combine ces deux paramètres pour déterminer le taux de croissance de nouvelles souches virales.
Le modèle montre que les variants qui engendrent rapidement de nouvelles infections secondaires ont un plus grand avantage de croissance par rapport aux souches existantes lorsque le virus se transmet plus facilement entre les individus et que l’épidémie se propage rapidement. A l’inverse, lorsqu’il y a moins de transmission et que l’épidémie décroit, les variants qui mettent plus de temps à engendrer de nouvelles infections ont un avantage. Par exemple, lorsque des mesures de contrôle comme le port de masque ou la distanciation sociale sont en place, un variant créant de nouvelles infections plus tardivement peut être avantagé.
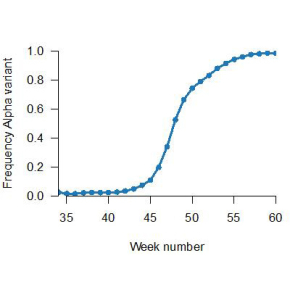
Les scientifiques ont appliqué leur modèle aux données des épidémies de variants Alpha et Delta au Royaume-Uni. Ils ont inféré qu’Alpha et Delta ne modifiaient pas le timing des infections secondaires par rapport aux souches précédemment en circulation. Mais le variant Alpha a un avantage de transmission estimé de 54 % par rapport aux souches précédentes et le variant Delta a un avantage de transmission de 140 % par rapport à Alpha.
Ces résultats suggèrent que le moment des infections secondaires et les taux de transmission jouent tous deux un rôle important sur la vitesse avec laquelle un virus se propage. Ce nouveau modèle mathématique pourra aider les épidémiologistes à mieux prédire la trajectoire des nouveaux variants de SARS-CoV-2 et à déterminer comment contrôler au mieux leur propagation.
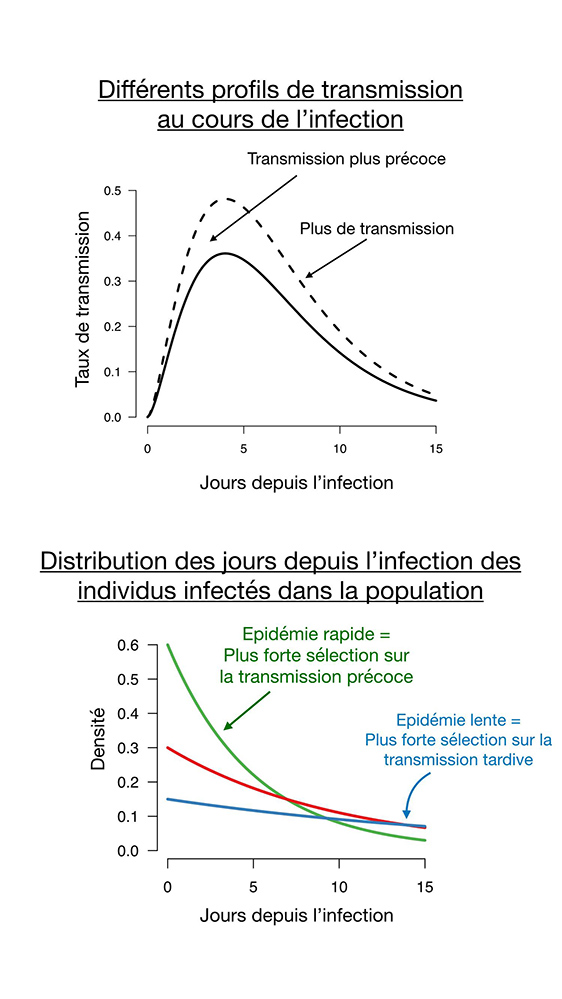
Figure : Pourquoi la sélection sur les variants dépend de la vitesse de l’épidémie.
En haut, différents profils de transmission sont illustrés. Le profil de transmission est le taux de transmission d’un individu infecté en fonction du temps depuis son infection, illustré en ligne continue pour les souches « historiques ». Il est typiquement maximum aux alentours de 5 jours après l’infection. Les variants modifient ce profil et peuvent conférer plus de transmission (ligne en tireté) ou bien une transmission plus précoce (ligne en pointillés).
Le panneau du bas présente les distributions des temps depuis l’infection chez les individus infectés de la population pour une épidémie rapide ou lente. Une épidémie rapide résulte en une majorité d’individus infectés récemment et une plus forte sélection sur la transmission précoce.
Pour en savoir plus :
Selection for infectivity profiles in slow and fast epidemics, and the rise of SARS-CoV-2 variants.
Blanquart F, Hozé N, Cowling BJ, Débarre F, Cauchemez S.
Elife. 19 mai 2022 . doi: 10.7554/eLife.75791.
Contact
Laboratoires
Centre interdisciplinaire de recherche en biologie - CIRB (CNRS/Inserm/Collège de France/PSL)
11 place Marcelin Berthelot
75005 Paris
Génomique évolutive, modélisation et santé - GEMS (Institut Pasteur/CNRS)
Institut Pasteur
28 rue du Docteur Roux
75724 PARIS CEDEX 15