Certains promoteurs ont une folle envie de se déméthyler avant l’heure…
La méthylation de l’ADN est une modification qui touche le plus souvent la cytosine, une des quatre bases de l’ADN. Elle est impliquée dans différents processus physiologiques et sa dérégulation est souvent associée au cancer. La déméthylation des gènes est corrélée à leur expression, dans le bon type cellulaire et au bon stade de développement. Les chercheurs ont montré que certains éléments de régulation d’un locus immunologique, normalement actifs aux stades tardifs du développement des lymphocytes B, sont déméthylés très tôt au cours du développement embryonnaire suggérant l’existence de mécanismes de marquage avant même l’apparition de la lignée lymphocytaire B. Cette étude est publiée dans la revue PLoS Genetics.
La méthylation de l’ADN est un mécanisme de régulation épigénétique commun chez les vertébrés. Elle est impliquée dans différents processus physiologiques au cours du développement et de la différenciation et sa dérégulation est souvent associée à des pathologies comme le cancer. Les cytosines de l’ADN sont méthylées de manière prédominante dans le contexte du dinucléotide CpG. La méthylation des CpG au niveau des régions promotrices est étroitement associée à une extinction transcriptionnelle. Les sites CpG déméthylés sont au contraire associés à des promoteurs actifs. Cela est moins évident au cours de la progression de la transcription et il a été montré dans plusieurs cas que les CpG intragéniques méthylés ne bloquent pas l’élongation transcriptionnelle. Ces données ont abouti à proposer que l’initiation de la transcription soit plus sensible à la méthylation des CpG.
Les chercheurs ont analysé les profils de méthylation de la région constante du locus des chaînes lourdes des immunoglobulines (IgH) qui s’exprime normalement aux stades tardifs du développement des lymphocytes B. Ils ont montré, en utilisant différentes lignées de souris que, contrairement au dogme qui prévaut actuellement, les profils de méthylation des éléments de régulation (promoteurs, enhancers et insulateurs) du locus IgH sont établis et maintenus indépendamment de l’activation des cellules B et de la transcription du locus. La progression transcriptionnelle à travers les régions constantes des gènes n’affecte nullement l’état hyperméthylé de leurs CpGs.
De manière surprenante, alors que les enhancers du locus sont déméthylés de manière restreinte aux lymphocytes B, deux promoteurs (notamment le promoteur Ig3) impliqués dans des réponses immunitaires adaptatives spécifiques, sont hypométhylés très tôt au cours de l’embryogenèse, i.e. à des stades antérieurs à l’apparition de la lignée B.
Par ailleurs, les promoteurs hypométhylés sont ciblés par différentes voies de déméthylation, incluant la voie controversée d’AID, tôt au cours du développement embryonnaire. Par contre, ces voies sont dispensables au maintien de l’état déméthylé de ces promoteurs dans la lignée B.
Ces données impliquent la méthylation de l’ADN dans le pré-marquage épigénétique du locus IgH plutôt que dans la régulation de son expression, et suggèrent que certaines réponses immunitaires adaptatives sont, en partie du moins, pré-programmées.
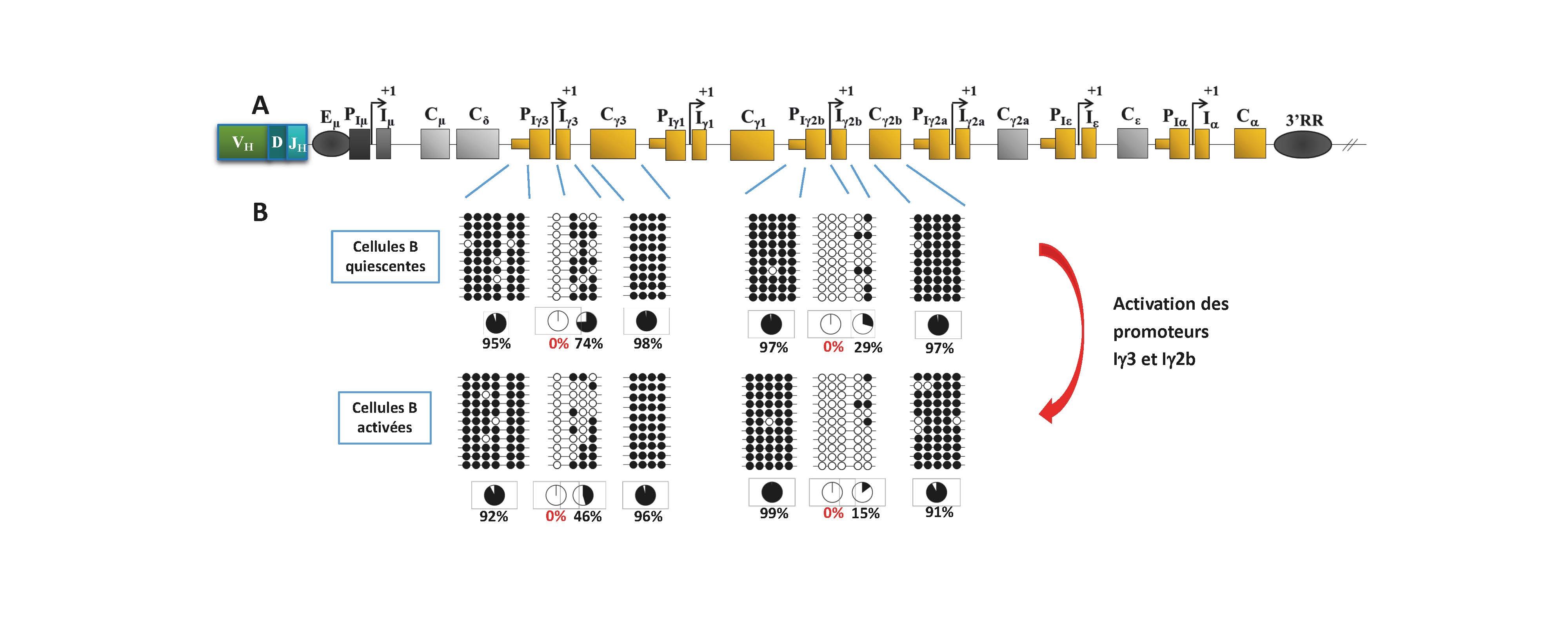
Figure. (A). Schéma du locus IgH murin réarrangé. Les gènes constants ont une structure similaire. Leur transcription est initiée à partir de promoteurs appelés I et est sous le contrôle d’un super-enhancer appelé 3’RR. (B). Illustration des profils de méthylation des CpG des gènes constants g3 et g2b. Les promoteurs PIg3 et PIg2b sont déméthylés (cercles blancs) avant comme après activation, et ce de manière très focalisée. +1 indique les sites d’initiation de la transcription. L’élongation transcriptionnelle à travers les régions constantes Cg3 et Cg2b n’induit aucun changement dans l’état de méthylation de ces régions qui demeurent hyperméthylées (cercles noirs).
En savoir plus
Developmental regulation of DNA cytosine demethylation at the immunoglobulin heavy chain constant locus
Oudinet C, Braikia FZ, Dauba A, Santos JM and Khamlichi AA
PLoS Genetics February 19, 2019 https://doi.org/10.1371/journal.pgen.1007930