Accéder à la face cachée des protéines neuronales grâce à l’expansion du code génétique
Les progrès de l’imagerie biologique sont intrinsèquement liés aux progrès des méthodes de marquage. Le développement de l’imagerie à super-résolution nécessite de nouvelles approches pour marquer les protéines cibles avec de petites sondes nanométriques. Cet article, publié dans la revue dans Nature Communication, décrit le développement d’un pipeline complet de marquage et d’imagerie utilisant l’expansion du code génétique et les acides aminés non naturels qui permet de marquer par fluorescence les épitopes masqués dans les protéines transmembranaires dans les neurones vivants.
Au cours des 15 dernières années, les progrès de la microscopie photonique à super-résolution ont révolutionné la façon dont les neuroscientifiques perçoivent les processus neuronaux-clés tels que l’organisation nanométrique des synapses et axones ou le trafic des protéines au niveau de la molécule unique. Les améliorations apportées aux différentes méthodes d’imagerie à super-résolution ont permis d’atteindre régulièrement des résolutions de l’ordre de ~20 nanomètres. Cependant, l’élucidation de l’organisation des protéines cibles à une résolution pratiquement moléculaire nécessite non seulement une grande résolution, mais également une densité et une spécificité de marquage élevées, ainsi qu’une distance entre le rapporteur fluorescent et la cible (erreur de liaison) considérablement plus petite que la résolution d’imagerie souhaitée. Les méthodes classiques de marquage utilisées pour l’imagerie de fluorescence telles que le marquage de la protéine-cible avec un complexe anticorps-fluorochrome ou la fusion génétique avec une protéine fluorescente rapportrice sont limitées dans leur utilisation, en particulier dans les neurones vivants. Il y a donc un besoin pressant de développer des méthodes d’étiquetage alternatives qui ne dépendent pas de l’accessibilité de l’épitope et avec des tailles compatibles avec la précision nanométrique de l’imagerie à super-résolution.
L’organisation à l’échelle nanométrique des synapses est un système modèle idéal pour l’application de méthodes innovantes d’imagerie et de marquage en raison de sa complexité et de sa diversité ainsi que du lien étroit entre l’organisation et la fonction des synapses. Le complexe des récepteurs AMPA (AMPAR) du glutamate est responsable de la transmission synaptique excitatrice. Dans ce complexe, la famille des protéines auxiliaires TARP sont des modulateurs clés de la transmission synaptique et de la plasticité médiées par les AMPAR. Elle sont caractérisées par deux boucles extracellulaires non accessibles au marquage par des anticorps du fait de la proximité des AMPAR. Ceci a considérablement entravé notre compréhension de leurs propriétés par un manque d’outils d’étiquetage et d’imagerie appropriés.
Le marquage chimique de type "click" repose sur l’utilisation de 2 groupes réagissant entre eux très spécifiquement, c’est-à-dire sans interférer notamment avec l’environnement biologique. Cette caractéristique (bioorthogonalité) permet de réaliser des couplages et marquages in vivo. Ainsi, l’expansion du code génétique (GCE) offre la possibilité d’incorporer des acides aminés non canoniques (ncAA) contenant des groupes bioorthogonaux dans une protéine cible. En remplaçant un codon natif à une position précise dans l’ADN qui code pour la protéine cible par un codon tel que le codon stop TAG, une protéine modifiée peut ensuite être exprimée dans les cellules hôtes souhaitées par l’ajout d’une amino acyltransférase (RS) modifiée et des ARN de transfert orthogonaux à la machine de traduction des protéines de l’hôte qui reconnaissent le codon correspondant UAG sur l’ARN codant pour cette protéine.
Dans le cadre d'une collaboration avec l'Université de Würzburg, les scientifiques ont développé un pipeline complet de marquage bioorthogonal par GCE qui permet pour la première fois de marquer des protéines dans des neurones vivants dans des cultures hippocampiques primaires et organotypiques. Ils ont pu ainsi marquer et visualiser les TARP de surface dans les neurones vivants sans perturbation de leur fonction. En utilisant cette approche, ils montrent la distribution subcellulaire différentielle des TARP g2 et g8 par microscopie optique et super-résolution. De plus, on peut voir qu’il n’y a pratiquement pas de TARP non associés à des AMPAR à la surface des neurones de l’hippocampe. Ce développement ouvre de nouvelles portes à l’étude des protéines difficiles à marquer dans les neurones vivants.
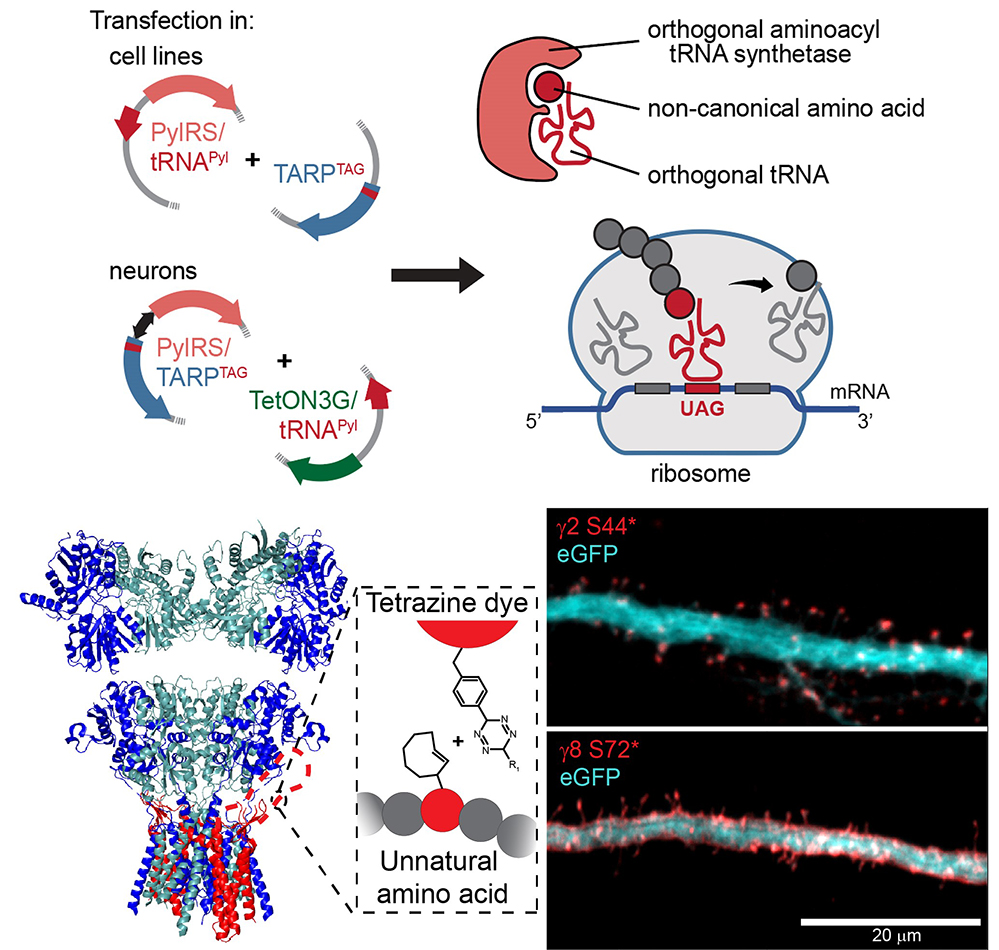
Figure : Haut : Stratégie d’expansion du code génétique (GCE) et insertion d’un acide aminé non naturel. Les plasmides codant pour une TARP mutée par insertion d’un codon stop, l'usage d'un ARN de transfert (tRNA) reconnaissant ce codon Stop et une ARN synthétase modifiée (PylRS) sont transfectés dans des cellules hétérologues ou des neurones, qui sont alors incubés avec un acide aminé non naturel qui s’incorpore au site du codon stop.
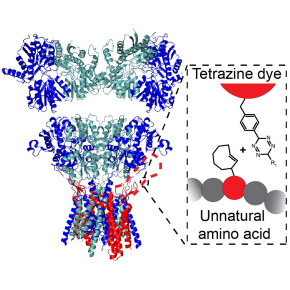
Bas : Structure cristallographique d’un récepteur AMPA tétramérique (gauche, sous-unités bleu et vertes) associé à deux protéines auxiliaires TARP g8 en rouge. La boucle extracellulaire en pointillé est non-structurée et donc non résolue au niveau structural. Son interaction étroite avec le récepteur AMPA empêche son marquage avec un anticorps ou son étiquetage avec une protéine fluorescente. En insérant un codon Stop TAG dans la séquence de g8 au niveau de cette boucle, et en fournissant aux neurones un acide aminé non naturel correspondant à la machinerie de traduction citée en haut, on peut faire incorporer cet acide aminé non naturel à la protéine et le marquer avec un fluorophore par chimie clic (tetrazine dye). A droite, on voit en rouge les TARP g2 et g8 dans deux dendrites exprimant également la GPF pour en révéler la forme. Notez la distribution distincte de g2 et g8.
Pour en savoir plus :
Bioorthogonal labeling of transmembrane proteins with non-canonical amino acids unveils masked epitopes in live neurons
Diogo Bessa-Neto, Gerti Beliu, Alexander Kuhlemann, Valeria Pecoraro, Sören Doose,
Natacha Retailleau, Nicolas Chevrier, David Perrais, Markus Sauer & Daniel Choquet
Nature Communications, 18 Novembre 2021. https://doi.org/10.1038/s41467-021-27025-w
Contact
laboratoire
Institut interdisciplinaire de neuroscience (CNRS et Université de Bordeaux) & Bordeaux Imaging Center (CNRS et Université de Bordeaux et INSERM)
Centre Broca Nouvelle Aquitaine, 146 rue Léo saignat,
33076 Bordeaux Cedex, France