Découverte d’une nouvelle structure de protéine permettant de mieux comprendre le métabolisme énergétique bactérien
Dans un article paru dans Nature Structural and Molecular Biology, des scientifiques décrivent un nouveau module structural au cœur du métabolisme énergétique bactérien. Cette découverte ouvre la voie à de nouvelles applications biotechnologiques.
L’exploration de la « matière noire microbienne » pour découvrir de nouvelles structures
Les avancées spectaculaires du séquençage et de l’analyse des génomes ont profondément bouleversé notre compréhension de la biodiversité microbienne. Ainsi, les analyses métagénomiques permettent de caractériser l’ensemble des gènes présents dans un échantillon environnemental, capturant la diversité génétique de toutes les espèces microbiennes présentes. Pourtant, près de 40 % des gènes issus des métagénomes environnementaux restent sans annotation fonctionnelle, c’est-à-dire que leur fonction biologique n’est pas encore définie. Cette "matière noire microbienne" représente un réservoir d’activités enzymatiques encore inexplorées, suscitant un vif intérêt pour la découverte de nouvelles fonctions.
Dans une étude parue dans Nature Structural and Molecular Biology, des scientifiques se sont intéressés à des enzymes, appelées oxydoréductases, indispensables à la conversion et au transfert d’énergie dans les cellules. Ces enzymes sont souvent des métalloprotéines qui contiennent un ou plusieurs ions métalliques essentiels à leur fonction catalytique, sont apparues aux origines de la vie sur Terre. Paradoxalement, malgré leur diversité, ces enzymes reposent en réalité sur un nombre limité de modules structuraux, dont l’inventaire est encore loin d’être complet.
Découverte d’un nouveau modèle structural
En combinant des approches de bioinformatique à des techniques de pointe de biologie structurale, les scientifiques ont pu identifier un nouveau module structurel : une étonnante structure tubulaire qui connecte des oxydoréductases à la membrane de la bactérie et ainsi à des quinones, des petites molécules membranaires qui sont de véritables connecteurs électrochimiques essentiels pour le transfert d’énergie dans les cellules. Ce modèle structural est décrit pour une métalloenzyme impliquée dans le métabolisme du carbone chez la bactérie du sol Bacillus subtilis, catalysant la conversion du formiate en dioxyde de carbone.
Les scientifiques mettent en évidence, à l’extrémité du tube, un domaine baptisé HMP (Helical Membrane Plug-in), qui assure une interaction transitoire avec la membrane. Cette découverte permet également d’attribuer une fonction à une famille de protéines jusqu’ici non caractérisée, présente dans plusieurs domaines du vivant. Le module contenant le domaine HMP pourrait n’être qu’un exemple parmi d’autres modules structuraux encore inconnus, jouant un rôle clé dans les interactions protéine-protéine ou entre compartiments cellulaires. Ces modules facilitent l’intégration des enzymes dans les réseaux métaboliques, renforçant ainsi l’adaptabilité des micro-organismes. Grâce à leur modularité, ces domaines pourraient aussi être exploités en biotechnologie pour concevoir des enzymes ancrées à des compartiments cellulaires spécifiques.
En résumé, cette étude révèle un mécanisme inédit d’ancrage des oxydoréductases à la membrane, au cœur du métabolisme énergétique microbien.
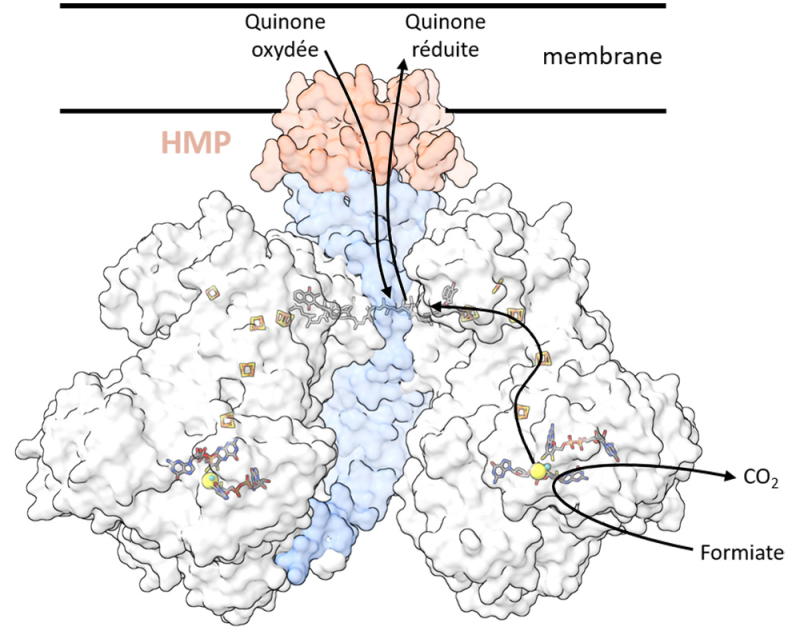
Figure : Organisation structurale du complexe formiate déshydrogénase chez la bactérie Bacillus subtilis révélée par cryo-microscopie électronique. Les sous-unités catalytiques (en gris) s’assemblent autour d’un tube central formé par quatre sous-unités entrelacées (en bleu) terminées par le domaine HMP (en orange), assurant le lien avec la membrane et les quinones. Les analyses bio-informatiques révèlent que ce type d’architecture est partagé par au moins trois autres familles d’oxydoréductases chez divers micro-organismes.
En savoir plus : Broc M, Cherrier MV, Uzel A, Arias-Cartin R, Arnoux P, Brasseur G, Seduk F, Guigliarelli B, Legrand P, Pierrel F, Schoehn G, Maté MJ, Martin L, Grimaldi S, Nicolet Y, Magalon A, Walburger A. A scaffold for quinone channeling between membrane and soluble bacterial oxidoreductases. Nat Struct Mol Biol. 2025 Aug 25. doi: 10.1038/s41594-025-01607-4. Epub ahead of print. PMID: 40855134.
Contact
Laboratoires
- Laboratoire de chimie bactérienne - LCB (CNRS/Aix-Marseille Université)
31 Chemin Joseph Aiguier
13009 Marseille - Institut de biologie structurale - IBS (CEA/CNRS/Université Grenoble Alpes)
71 Avenue des Martyrs
38000 Grenoble
