Décryptage du code permettant à l’auxine, une hormone végétale, d’orchestrer le développement des plantes
L’auxine, une hormone végétale, agit comme un signal qui joue le chef d’orchestre pour contrôler l’expression des gènes au cours du développement des plantes. Dans un article publié dans Cell, des scientifiques ont pu décrypter le code cellulaire permettant à l’auxine de contrôler cette expression des gènes. Ils montrent que ce code repose sur une interaction combinatoire entre les nombreuses protéines qui médient la réponse à l’auxine et les différents motifs de l’ADN sur lesquels elles se lient.
L’Auxine, une molécule signal indispensable au développement de la plante
Les organismes multicellulaires, qu’ils soient végétaux ou animaux, utilisent un petit nombre de molécules qui, telles des signaux, peuvent réguler leur développement et assurer la construction de l’organisme. Comprendre comment ces signaux développementaux agissent sur le génome pour réguler l’expression des gènes dans l’espace et le temps est fondamental. Un même signal peut activer différents gènes dans différentes parties de l’organisme, mais l’orchestration de ces mécanismes reste encore énigmatique.
L’auxine, hormone essentielle impliquée dans toutes les phases de développement des plantes, est un parfait exemple de la manière dont un même signal peut être utilisé pour générer une multiplicité de profils d’expression de gènes au cours du développement.
L’Auxine et les « Auxin Response factors » (ARF), un système complexe
La régulation de l’expression des gènes en réponse à l’auxine est médiée par une famille de facteurs de transcription, les Auxin Response Factors (ARF), qui sont au nombre de 23 chez l’Arabette. Ces facteurs sont capables de se lier à l’ADN et réguler l’expression des gènes en activant ou inhibant la synthèse des ARN messagers (la transcription) qui pourront ensuite être traduits en protéines. Il est communément admis que ces ARF peuvent être soit des activateurs, soit des répresseurs de l’expression des gènes. Leur nombre et le fait que certains soient des activateurs et d’autres des répresseurs, ont amené à l’hypothèse qu’il pourrait y avoir une compétition entre ces ARF. Cette compétition, liée à la présence de cocktails d’ARF différents dans les cellules, expliquerait la capacité de l’auxine à générer différents profils d’expression au cours du développement des plantes. Les ARF établiraient ainsi un code combinatoire. Mais la multiplicité des ARF chez les plantes à fleurs rendait très difficile la validation de cette hypothèse.
Décryptage du code transcriptionnel de l’Auxine
Dans un article publié dans la revue Cell, des scientifiques, en utilisant la plante modèle Arabette, ont maintenant réussi à décrypter ce code. Pour obtenir ces résultats, ils ont construit des promoteurs synthétiques, c’est-à-dire des séquences d’ADN artificielles qui contrôlent l’expression des gènes en réponse à l’auxine. Ces promoteurs possèdent des motifs de liaisons qui sont reconnus par différents types d’ARF, qu’ils soient activateurs ou répresseurs. Ils ont ainsi créé un environnement simplifié de régulation de l’expression de gènes par les ARF. En testant ces promoteurs dans divers systèmes biologiques de complexité croissante, ils ont découvert que chaque ARF a une préférence pour certains promoteurs. Plus surprenant encore, l’effet d’un ARF, activateur ou répresseur, peut changer en fonction des sites de liaison présents dans le promoteur. Cela signifie que l’activité d’un ARF n’est pas figée, mais dépend de son interaction avec les motifs spécifiques du promoteur, révélant ainsi un code transcriptionnel bi-couche : le type d’ARF et le motif de liaison.
L’ensemble de ces premiers résultats suggéraient que les différents promoteurs synthétiques devaient conduire à des profils d’expression spatiaux distincts. Pour le vérifier, les scientifiques ont utilisé ces promoteurs synthétiques pour exprimer des protéines fluorescentes dans la racine de l’Arabette. Ils ont montré que ce code bi-couche façonne bien l’expression des gènes en réponse à l’auxine. En utilisant des techniques avancées de séquençage d’ARN, qui permettent de quantifier les ARN messagers (ceux qui seront traduits en protéine) dans chaque cellule, ils ont montré que la présence des différents motifs de liaison des ARF dans le promoteur de gènes régulés par l’auxine contribue à la diversité des profils d’expression. Cela signifie que le code bi-couche généré par les ARF joue un rôle clé dans la diversification des réponses à l’auxine dans les tissus végétaux.
En combinant différents motifs de liaison des ARF dans une série de nouveaux promoteurs synthétiques, les scientifiques ont pu encore diversifier les profils d'expression observés dans la racine de l’Arabette. Cette expérience démontre la capacité du code transcriptionnel à deux niveaux à générer des profils d'expression spatiaux variés, qui ne sont pas simplement le reflet de la distribution de l'auxine dans la plante. Grâce à cette étude, il devient possible de comprendre comment un même signal de développement peut induire une multitude de réponses transcriptionnelles, malgré la complexité des interactions moléculaires en jeu. Cette avancée ouvre de nouvelles perspectives pour l'agriculture et la médecine, en permettant de mieux contrôler et prédire les réponses des organismes aux signaux de développement.
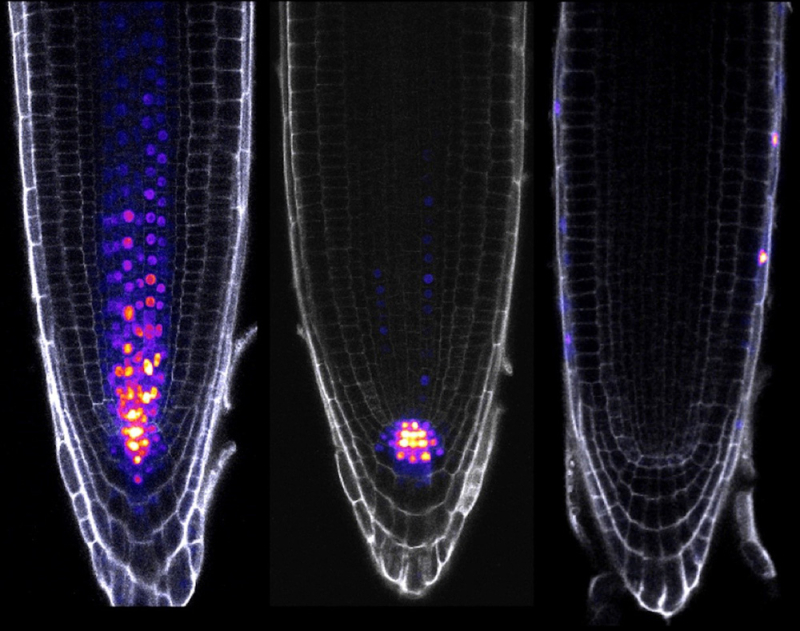
Figure : Trois exemples d’expression dans la racine de l’Arabette de trois rapporteurs synthétiques construits à partir de trois différents motifs de liaison des ARF et contrôlant l’expression de la protéine fluorescente mTurquoise. Images prises par microscopie confocale dans le plan médian de la racine. Les parois des cellules sont visualisées en gris. La fluorescence de la mTurquoise est visualisée selon une gamme de couleurs allant du violet au jaune-orange.
Référence : Synthetic deconvolution of an auxin-dependent transcriptional code. Raquel Martin-Arevalillo, Bruno Guillotin, Jonas Schön, Alice Hugues, Marie-France Gerentes, Kun Tang, Jérémy Lucas, Emmanuel Thévenon, Marianne Dreuillet, Graeme Vissers, Mohammed Mohammed Ateequr, Carlos S. Galvan-Ampudia, Guillaume Cerutti, Jonathan Legrand, Coralie Cance, Annick Dubois, François Parcy, Kenneth D. Birnbaum, Matias D. Zurbriggen, Renaud Dumas, François Roudier and Teva Vernoux.
Cell, 15 avril 2025, DOI : 10.1016/j.cell.2025.03.028
Contact
Laboratoire
Laboratoire Reproduction et Développement des Plantes - RDP (CNRS/INRAE/ENS Lyon)
Site Monod
46 allée d’Italie
69007 Lyon - FRANCE