La découverte d’un nouveau mécanisme moléculaire doit permettre de mieux cibler les protéines impliquées dans l’insuffisance cardiaque
Dans l’insuffisance cardiaque, le cœur ne parvient plus à alimenter correctement l’organisme en sang, ce qui affecte durement la qualité de la vie. La protéine EPAC1 est une cible thérapeutique de premier plan pour prévenir et soigner cette maladie. Dans cette étude publiée dans Nature communications, les scientifiques ont découvert qu’elle possède un mécanisme moléculaire inattendu, qui ouvre de nouvelles perspectives pour la découverte et l’amélioration de médicaments.
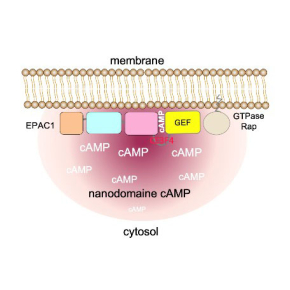
EPAC1 est un facteur d’échange nucléotidique (ou GEF) des petites GTPases Rap, c’est-à-dire qu’il les active en stimulant l’échange du nucléotide GDP par du GTP. Ce régulateur est est strictement régulé par l'AMP cyclique (AMPc), une petite molécule dite « second messager » qui propage à l’intérieur des cellules les signaux émis par des hormones ou des neurotransmetteurs. En l’absence d’AMPc, le site catalytique d’EPAC1 est obstrué par des interactions intramoléculaires qui le rendent complètement inerte. Lorsque la concentration en AMPc augmente en réponse à un signal extracellulaire, EPAC1 réorganise sa structure tridimensionnelle, révèlant son site actif et permettant l’activation des GTPases Rap.
Pendant longtemps, il était accepté que la fixation de l’AMPc à EPAC1 se produisait dans le cytosol, provoquant ensuite sa translocation vers les membranes où se trouvent les GTPases Rap actives. Une découverte récente a remis ce modèle en question : en effet l’AMPc ne diffuse pas librement comme on le croyait, ce qui permettrait à sa concentration d’augmenter rapidement dans le cytosol. Au contraire, l’AMPc est compartimentée dans des nanodomaines proches de la membrane plasmique, à proximité des récepteurs des hormones et des neurotransmetteurs. Par conséquent, sa concentration reste faible dans le cytosol. Or on sait depuis longtemps qu’EPAC1 a une affinité très faible pour l’AMPc en solution, impliquant qu’EPAC1 n’a pas les caractéristiques pour être activé par l’AMPc dans le cytosol.
L’étude publiée dans Nature Communications résout cette incohérence en révélant que la membrane plasmique est un composant essentiel de l'activation d'EPAC1, passé inaperçu jusqu’ici. Au moyen de protéines purifiées et de membranes artificielles imitant la membrane plasmique, les scientifiques ont montré que ces membranes activent EPAC1 indépendamment de l'AMPc, avec une efficacité du même ordre. De façon remarquable, les membranes augmentent l’affinité d’EPAC1 pour l'AMPc de plus de deux ordres de grandeur, passant du micromolaire au nanomolaire. L’activité maximale d’EPAC1 est finalement obtenue par l’action en synergie de l’AMPc et des membranes. Ces résultats permettent d’élaborer un nouveau modèle de l’activation d’EPAC1 qui rend compte de l’ensemble des observations. Dans ce modèle, l’activation d’EPAC1 dans les cellules est amorcée par son interaction préalable avec les membranes. En association avec la membrane, l’affinité d’EPAC1 pour l’AMPc augmente considérablement, ce qui permet son activation maximale par l’AMPc au voisinage de la membrane en réponse à un signal extracellulaire. Inversement, le complexe EPAC1-cAMP soluble est un intermédiaire réactionnel négligeable à la concentration cytosolique de l’AMPc. Ce modèle a pour corollaire qu’EPAC1 possède les caractéristiques pour fonctionner au sein de nanodomaines d’AMPc proches de la membrane, ouvrant ainsi la voie à de nouvelles recherches.
Cette découverte a des implications étonnantes pour la découverte et l’amélioration d’inhibiteurs d’EPAC1. Les scientifiques ont revisité l’action de CE3F4, un composé chimique prometteur qui inhibe l’activation des GTPases Rap par EPAC1 in vitro et en cellules et qui prévient l’arythmie cardiaque chez le rat et la souris. Ils montrent qu’à concentration physiologique d’AMPc, CE3F4 n’inhibe EPAC1 qu’à condition qu’EPAC1 soit complètement activé par à la fois les membranes et l'AMPc. Autrement dit, EPAC1 a besoin d’être activé pour pouvoir être inhibé par CE3F4. Cette découverte réoriente désormais la stratégie d’amélioration du composé.
Cette étude reformule les concepts antérieurs de la signalisation de l'AMPc passant par les petites GTPases, révélant un rôle primordial des membranes avec des implications importantes pour la découverte de médicaments pour combattre l’insuffisance cardiaque. Elle illustre aussi la pertinence de la biologie structurale intégrative, combinant ici des reconstitutions en membrane, de la cristallographie, du SAXS et de l’échange hydrogène/deutérium couplé à la spectrométrie de masse, pour élucider des questions biologiques fondamentales.
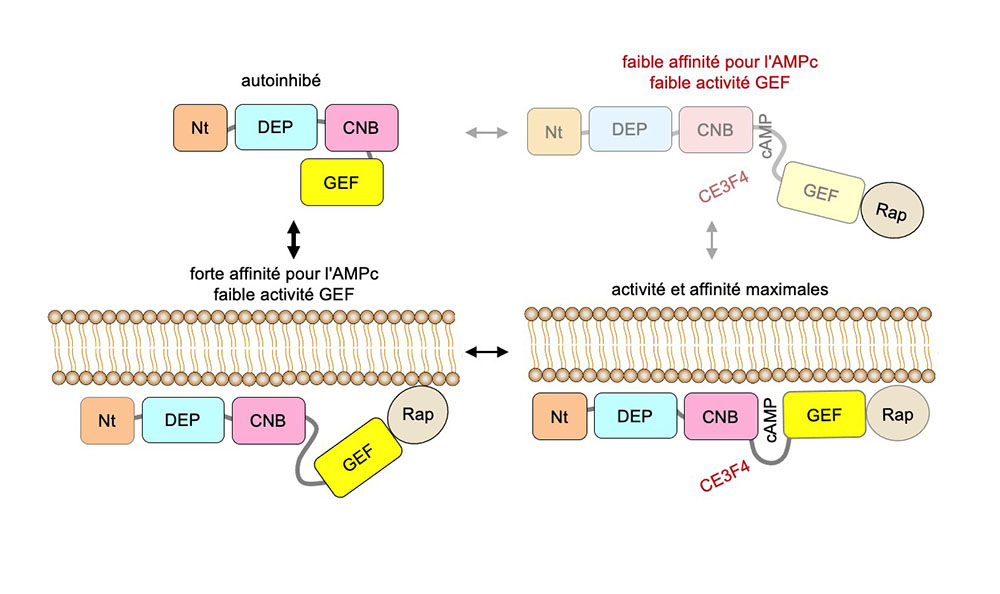
Figure : EPAC1, le facteur d’échange de la petite GTPases Rap, est activé par l’AMPc et la membrane plasmique. Son activation nécessite des réarrangements importants de ses 4 domaines (indiqués en orange, bleu, rose et jaune). L’état intermédiaire activé par l’AMPc est négligeable dans le cytosol, où la concentration de l’AMPc et l’affinité d’EPAC1 pour l’AMPc sont faibles. EPAC1 est de ce fait pré-activé par son interaction avec la membrane, où il active la petite GTPase Rap, elle-même localisée à la membrane, en réponse à un signal AMPc. EPAC1 possède ainsi les caractéristiques pour être activé au sein de nanodomaines d’AMPc à la périphérie de la membrane. Ce mécanisme conduit à revisiter le mécanisme d’action de CE3F4, un inhibiteur prometteur dans l’insuffisance cardiaque.
En savoir plus :
Membranes prime the RapGEF EPAC1 to transduce cAMP signaling. Sartre C, Peurois F, Ley M, Kryszke MH, Zhang W, Courilleau D, Fischmeister R, Ambroise Y, Zeghouf M, Cianferani S, Ferrandez Y, Cherfils J. Nat Commun. 2023 Jul 12;14(1):4157. doi: 10.1038/s41467-023-39894-4.
Contact
Laboratoire
Laboratoire de biologie et pharmacologie appliquée – LBPA (CNRS/Université Paris-Saclay)
Ecole Normale Supérieure Paris-Saclay
4 avenue des Science,
91190 Gif-sur-Yvette