Surveillance des ARN dans le troisième domaine du vivant
Les archées, constituant l'un des trois domaines du vivant, sont désormais considérées comme des modèles incontournables pour étudier les mécanismes moléculaires de la cellule et pour comprendre l'histoire évolutive. Ces résultats, publiés dans la revue Nucleic Acid Research, ouvrent de nouvelles perspectives sur les acteurs moléculaires en charge de la surveillance des ARN dans la cellule et donc de la régulation de l’expression des gènes.
Il y a plus de 40 ans que les archées (ou Archaea) ont été proposées comme un nouveau domaine du vivant au côté des bactéries et des eucaryotes. Au fil des ans, notre vision des micro-organismes archéens a évolué, passant d'extrémophiles microbiens exotiques à des organismes d'importance universelle utilisés pour élucider des questions biologiques fondamentales. Il est désormais acquis que ces organismes constituent de fait un groupe à part entière, unique par de nombreux aspects cellulaires et moléculaires, et seraient, dans l’arbre du vivant, une branche à l'origine du groupe des Eucaryotes.
Les archées sont omniprésentes dans tous les sites terrestres (> 20 % de la biomasse microbienne marine) et sont notamment présentes dans le microbiote humain. Pour comprendre leur impact sur l'environnement et la santé humaine, il est donc impératif de décoder leurs particularités à un niveau moléculaire. Les machineries moléculaires décryptées chez les archées fourniront des paradigmes clés pour comprendre les processus biologiques fondamentaux conservés dans le vivant. En particulier, les bases moléculaires de la régulation de l'expression des gènes, par des mécanismes post-transcriptionnels, sont encore mal connues. Un réseau d'hélicases à ARN, d'endoribonucléases et d'exoribonucléases régule la quantité et la qualité des ARN de toute cellule. Jusqu'à présent, les études mécanistiques se sont concentrées sur les systèmes bactériens et eucaryotes en raison du défi que représente l'identification des principaux acteurs de la maturation et la dégradation de l'ARN chez les Archées, car peu d’outils génétiques sont disponibles.
Les chercheurs ont choisi comme modèle d’étude des archées hyperthermophiles (Thermococales) et utilisé des approches génétiques, protéomiques et phylogénomiques. Les données assemblées constituent un jalon dans le domaine du métabolisme de l'ARN chez les archées. Conjointement aux précédents résultats montrant que les ribonucléases (RNases) de la famille des β-CASP, omniprésentes chez les archées, sont des acteurs clés du métabolisme de l'ARN, les données collectées soutiennent un modèle selon lequel la ribonucléase aRNase J (dont on ne retrouve l'homologue que chez les bactéries), l'hélicase à ARN de type Ski2 et l'exosome à ARN (dont les homologues sont eucaryotes) sont en interaction dans la plupart des cellules d'archées étudiées. Les données confirment que la ribonucléase aRNase J exerce potentiellement un contrôle sur la surveillance des ARN, à proximité du ribosome. Ces résultats suggèrent, pour la première fois, le rôle fondamental d’un complexe ribonucléase β-CASP / helicase à ARN dans le métabolisme de l'ARN chez les archées et soulignent l'aspect mosaïque des machines moléculaires où sont retrouvés associés à la fois des homologues bactériens et eucaryotes. Ces travaux ouvrent des perspectives sur l’évolution des acteurs clés au cœur de la régulation post-transcriptionnelle des gènes.
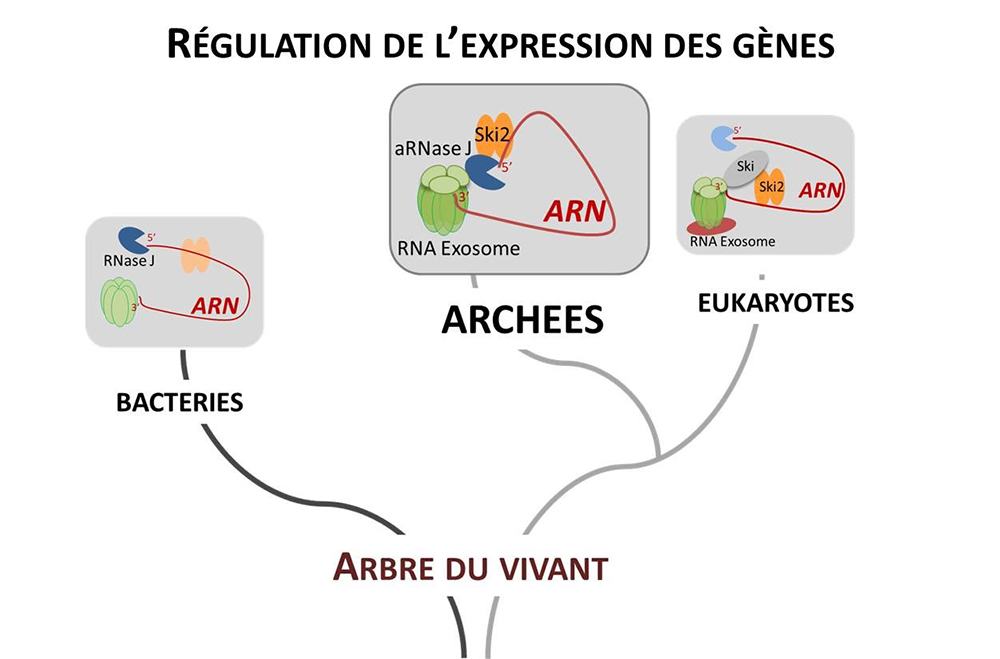
Figure : Représentation schématique de l’arbre du vivant avec les bactéries, les archées et les eucaryotes. Les machines moléculaires (en vert) et enzymes (exo-ribonucléases en bleu/ hélicases à ARN en orange), en charge de la dégradation des ARN, retrouvés en commun dans les trois branches du vivant sont représentés. Ces enzymes et machines sont les acteurs clés des voies métaboliques gérant la quantité et la qualité des ARN et sont donc fondamentaux dans la régulation de l’expression des gènes dans la cellule.
En savoir plus
RNA processing machineries in Archaea: the 5'-3' exoribonuclease aRNase J of the β-CASP family is engaged specifically with the helicase ASH-Ski2 and the 3'-5' exoribonucleolytic RNA exosome machinery.
Phung DK, Etienne C, Batista M, Langendijk-Genevaux P, Moalic Y, Laurent S, Liuu S, Morales V, Jebbar M, Fichant G, Bouvier M, Flament D, Clouet-d'Orval B.
Nucleic Acids Res. 2020 Feb 7. pii: gkaa052. doi: 10.1093/nar/gkaa052. [Epub ahead of print]
Contact
Laboratoire
Laboratoire de microbiologie et génétique moléculaire (LMGM) - (CNRS / Université Toulouse Paul Sabatier)
Bât. IBCG, 118, route de Narbonne
31062 TOULOUSE cedex 9, France