Une approche multi-omique pour décrypter un cancer du cerveau chez l’enfant
Le médulloblastome est la tumeur cérébrale maligne la plus fréquente chez l’enfant. Malgré des progrès considérables ces vingt dernières années, une fraction importante de ces tumeurs reste encore mal caractérisée. L’étude publiée dans la revue Cancer Cell décrit comment une analyse multi-omique (génome, transcriptome et protéome) a permis une meilleure caractérisation des médulloblastomes.
Le médulloblastome est la tumeur maligne du cerveau la plus répandue chez l’enfant. Cette tumeur se développe au niveau du cervelet avec un pic d’incidence entre 2 et 8 ans. Les progrès importants de ces dernières années ont permis d’améliorer considérablement la survie des patients. Aujourd’hui le taux de survie à 5 ans est de 80%. Malheureusement, ces résultats sont amenuisés par une toxicité importante des traitements (résection chirurgicale, chimiothérapie et radiothérapie).
Le médulloblastome est un cancer complexe. En se basant notamment sur des analyses transcriptomiques, il est aujourd’hui admis qu'il est divisé en quatre sous-groupes (Wingless, Sonic Hedgehog, Groupe 3 et Groupe 4). L’appartenance à ces différents sous-groupes est extrêmement importante puisqu’au-delà de signatures moléculaires distinctes, elle conditionne le pronostic de survie des patients. Ainsi, une meilleure connaissance de ces tumeurs apparaît essentielle pour le développement de stratégies thérapeutiques ciblées pour chacun des sous-groupes de médulloblastome.
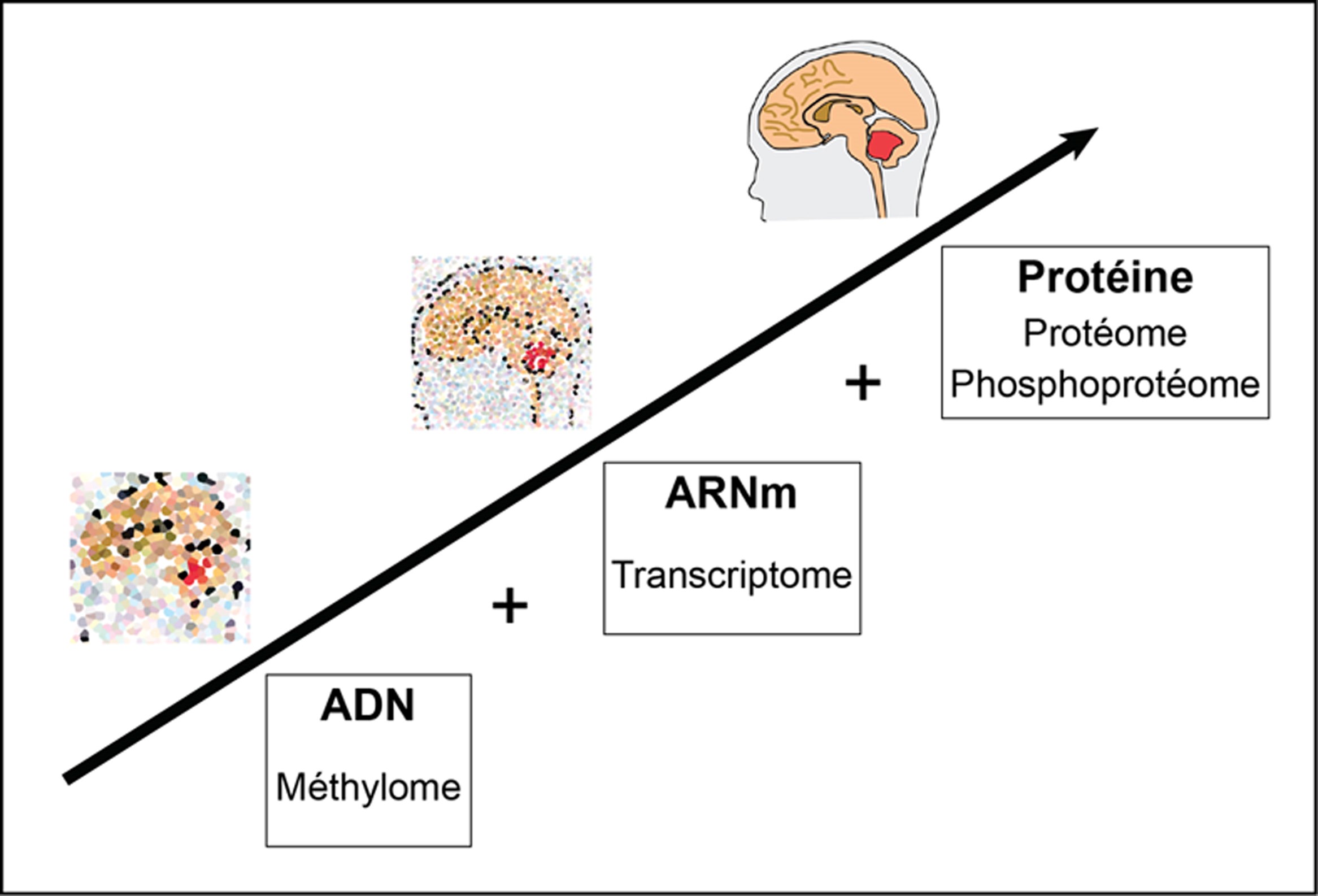
Dans le but de mieux caractériser les mécanismes impliqués dans la biologie du médulloblastome, les chercheurs ont réalisé une étude qui a consisté à décrypter le médulloblastome par une approche multi-omique intégrant la génomique, la transcriptomique et la protéomique (Méthylation du génome, séquençage de l’ARNm, analyse du protéome et du phosphoprotéome par spectrométrie de masse).
Pour cette étude, des échantillons de médulloblastome provenant de l’ensemble des groupes, ont été collectés en collaboration avec le service de neurochirurgie pédiatrique de l’hôpital Necker (Pr. S. Puget) ainsi que l’hôpital SickKids à Toronto (Dr. M. Taylor). A la suite de l’acquisition des données, une analyse bioinformatique intégrative a été réalisée pour déterminer si le protéome/phosphoprotéome était capable de révéler la présence d’acteurs et/ou de voies de signalisation spécifiquement enrichis à l’échelle protéique dans un ou plusieurs groupes, non détectés précédemment et qui pourraient être impliqués dans la formation et/ou la maintenance tumorale.
Les chercheurs ont démontré que la protéomique permet de déterminer le sous-groupe du patient. Ils ont également mis en évidence des voies de signalisation uniquement visibles au niveau protéique: pour la première fois, une activation de la voie des récepteurs à tyrosine kinase (RTK) a été révélée spécifiquement dans le Groupe 4 des médulloblastomes via l'activation des deux protéines ERBB4 et SRC.
En conclusion, grâce à cette approche multi-omique, les chercheurs ont pu apporter une meilleure connaissance de cette tumeur pédiatrique permettant d’entrevoir la possibilité de nouvelles stratégies thérapeutiques pour ce groupe le plus répandu de médulloblastomes.
© Antoine Forget
Pour en savoir plus :
Aberrant ERBB4-SRC Signaling as a Hallmark of Group 4 Medulloblastoma Revealed by Integrative Phosphoproteomic Profiling.
Forget A, Martignetti L, Puget S, Calzone L, Brabetz S, Picard D, Montagud A, Liva S, Sta A, Dingli F, Arras G, Rivera J, Loew D, Besnard A, Lacombe J, Pagès M, Varlet P, Dufour C, Yu H, Mercier AL, Indersie E, Chivet A, Leboucher S, Sieber L, Beccaria K, Gombert M, Meyer FD, Qin N, Bartl J, Chavez L, Okonechnikov K, Sharma T, Thatikonda V, Bourdeaut F, Pouponnot C, Ramaswamy V, Korshunov A, Borkhardt A, Reifenberger G, Poullet P, Taylor MD, Kool M, Pfister SM, Kawauchi D, Barillot E, Remke M, Ayrault O.
Cancer Cell. 2018 Sep 10;34(3):379-395.e7. doi: 10.1016/j.ccell.2018.08.002.