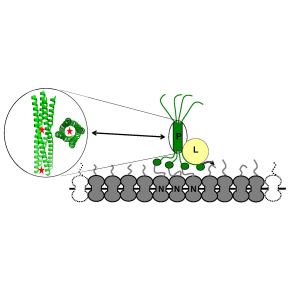
Un "exosquelette" protéique essentiel à l’activité de la polymérase du virus de la rougeole
La machinerie de réplication de virus de la rougeole est complexe. Des investigations fonctionnelles et structurales couplées à la modélisation apportent un nouvel éclairage sur l’interaction entre deux protéines du complexe polymérase : la protéine P stabilise la polymérase à la manière d’un exosquelette dynamique via la cohésion d’un cœur d’hélices surenroulées. L’étude, parue dans Science Advances, permettra la conception d’antiviraux et la compréhension d’autres mécanismes biologiques.
La propriété essentielle des virus est leur capacité à recopier leur matériel génétique et à le transmettre à d’autres cellules hôtes. Pour ce faire, ils doivent produire des ARN messagers traduits en protéines par la machinerie de traduction de l’hôte. Ils doivent également recopier fidèlement leur génome pour assurer leur descendance en nouveaux virus à transmettre à un autre hôte. Chez le virus de la rougeole, un membre de la famille des Paramyxoviridae, le génome est un ARN simple brin (de polarité négative). Ce génome a une double fonction matricielle. D’une part il sert à synthétiser chacun des transcrits viraux à partir desquels seront traduits les protéines virales. D’autre part, et dans une seconde étape, ce génome sert de matrice pour synthétiser une copie ARN complète du génome (de polarité positive) qui sera à son tour à l’origine de nouvelles particules virales infectieuses.
L’enzyme responsable de la synthèse des ARN viraux est la polymérase, ou protéine L, une protéine de grande taille qui possède tous les domaines enzymatiques nécessaires à la synthèse et la maturation des ARN viraux. La production de cette polymérase est délicate car il faut que la chaîne polypeptidique se replie correctement, et que les domaines enzymatiques soient aussi correctement orientés les uns par rapport aux autres. Intervient alors un « cofacteur », la protéine P, qui s’associe étroitement à la protéine L dès le début de la traduction de cette dernière. Elle la « soutient » ainsi au fur et à mesure de sa synthèse à la manière d’un exosquelette dynamique pour permettre son repliement ordonné et correct. L’ARN génomique du virus de la rougeole est recouvert par un homopolymère de protéines N, formant ainsi une nucléocapside hélicoïdale. Le génome étant enfoui dans cette structure, P assure la fixation de la polymérase L sur la nucléocapside via son domaine C-terminal (PXD) qui se lie à l’extrémité C-terminale de la protéine N (NTAIL). Cette extrémité est très souple et assimilable à des poils hérissés au-dessus d’une chenille dont le corps serait la partie relativement rigide de la nucléocapside.
Quatre chaines de P s’associent entre elles pour former un homo-tétramère qui est la forme active de ce cofacteur. La tétramérisation de P repose sur l’auto-assemblage d’une hélice alpha centrale pour former une « hélice d’hélices » ou hélice surenroulée (« coiled coil » en anglais) à la manière d’une corde torsadée ou toron. De part et d’autre de ce cœur tétramérique, émergent de longues séquences très souples, sans structure régulière (dites intrinsèquement désordonnées). La région désordonnée en aval du cœur tétramérique se termine par le domaine globulaire PXD assurant l’ancrage de P sur la nucléocapside.
En combinant des approches biochimiques, moléculaires, cellulaires, de biologie structurale et de modélisation, les chercheurs ont établi une cartographie plus fine du site de fixation de la L sur la P, et ont découvert le rôle critique de la partie C-terminale de l’hélice surenroulée dans le maintien de la L dans une conformation stable. De plus, ils ont découvert que pour assurer la capacité de L à synthétiser un ARN viral, la P requiert (i) la présence et la position d’une irrégularité du pas d’hélice dans le cœur surenroulé entraînant la formation d’un coude et (ii) une force de cohésion très précise entre les quatre hélices alpha de ce coeur (voir schéma inférieur de la figure). De manière remarquable, les structures des protéines P et L sont semblables chez les autres Paramyxoviridae responsables de nombreuses pathologies animales et/ou humaines (maladie de Carré du chien, encéphalite à virus Nipah, bronchiolite, oreillons … ). Il est donc logique de supposer que leurs protéines P et L interagissent de manière similaire. Ainsi, le maintien de l’interaction L-P constituerait une contrainte majeure à laquelle ce couple protéique aurait été sujet au cours de l’évolution afin de conserver une activité optimale pour la multiplication de ces virus. Ce mode complexe d’interaction entre deux protéines constitue d’une part une nouvelle cible moléculaire potentielle pour concevoir des antiviraux et, d’autre part, élargit la panoplie des fonctions biologiques assurées par les structures en hélices alpha surenroulées dans le monde du vivant.
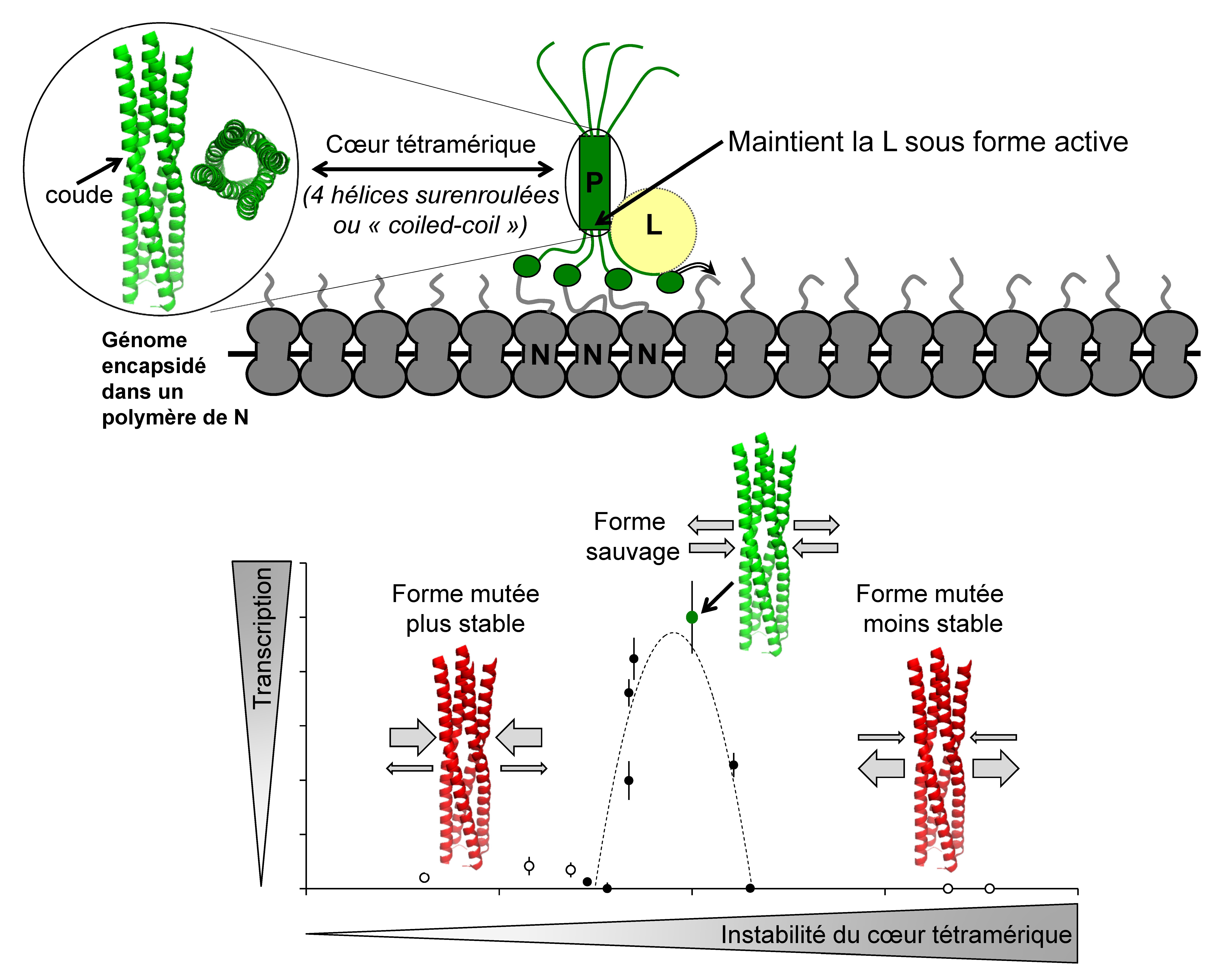
Schéma inférieur : L’efficacité avec laquelle la polymérase transcrit le génome viral en ARNm dépend du niveau de cohésion entre les quatre hélices surenroulées du cœur de la protéine P : seule une cohésion optimale (en haut, vert) permet la transcription par la L ; une cohésion trop forte (à gauche, rouge) ou trop faible (à droite, rouge) n’est pas compatible avec cette activité.
© Sonia Longhi & Denis Gerlier
Pour en savoir plus :
Regulation of measles virus gene expression by P protein coiled coil properties
Bloyet, L.M., Schramm, A., Lazert, Raynal, C.B., Hologne, M., Walker, O., Longhi,S. Gerlier, D.
Science Advances; 8 May Vol. 5, No. 5 DOI: 10.1126/sciadv.aaw3702