Des pincettes en ADN pour étudier l’interaction entre une médicament et sa cible à l’échelle molécule-unique
L’efficacité d’un médicament est fortement liée au temps que la molécule médicamenteuse passe accolée à sa cible, typiquement une protéine. Bien souvent si l’interaction est de longue durée la drogue aura un effet plus fort que si l’interaction est de courte durée. Une équipe pluridisciplinaire vient de décrire dans la revue Nature Nanotechnology une nouvelle approche à très haute résolution permettant d’observer, en temps réel, l’interaction d’une seule molécule médicamenteuse avec une seule molécule de cible. Cette observation du « quantum » d’interaction moléculaire ouvre de nouvelles perspectives dans le développement des médicaments mais aussi des anticorps thérapeutiques et de la science des matériaux.
L’ADN est une molécule chimique dotée de propriétés physiques uniques et qui dépassent de loin son rôle comme simple support de l’information génétique : elle est stable, inerte, facile à fabriquer, rigide à l’échelle de 50 nanomètres, et adressable via la séquence de bases qui la constitue (les fameuses « lettres » du code génétique A, T, G et C). D’ailleurs depuis plusieurs années maintenant les scientifiques exploitent ces propriétés en se servant de ce biopolymère comme nanomatériau pouvant être replié en formes arbitraires et fonctionnelles : il s’agit des fameux « origamis » en ADN.
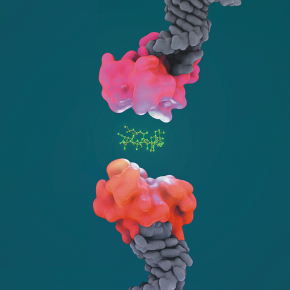
Inspirée par ces origamis, les chercheurs viennent de décrire une nouvelle façon d’assembler l’ADN permettant de s’en servir comme une pincette pour étudier les interactions entre molécules d’intérêt. Dans cette stratégie, ces dernières sont chimiquement couplées à deux endroits distincts le long de la pincette d’ADN qui est ensuite accrochée par une extrémité à une lame de verre traitée, et par l’autre extrémité à une microbille magnétique. La microbille est observée et manipulée grâce à un « piège magnétique », c’est-à-dire un microscope optique incorporant une paire d’aimants située au-dessus de la lame de verre et permettant d’appliquer à l’ADN une force de traction contrôlée, ceci via la bille magnétique. La position de la bille au-dessus de la lame de verre peut être mesurée par microscopie optique, en temps réel et avec une précision nanométrique.
Si les molécules se trouvent en interaction, la pincette est refermée et la bille est proche de la lame. Lorsque les molécules se dissocient la pincette s’ouvre et la bille s’éloigne de la lame. L’état de la pincette, refermée ou ouverte, est enregistré en temps réel. Le temps que la pincette passe dans l’état refermé avant de s’ouvrir représente ainsi la durée de l’interaction entre les molécules d’intérêt, et peut alors être déterminé avec une très grande précision et ceci pour chaque couple de molécules pris individuellement. La variation de cette durée en fonction de divers paramètres (intensité et direction de la force appliquée, température,…) permet une nouvelle caractérisation, chimique mais aussi physique, de l’interaction entre une molécule médicamenteuse et sa cible.
Afin de démontrer l’intérêt de l’approche, les chercheurs ont utilisé cette pincette moléculaire pour étudier l’interaction de la rapamycine avec sa cible thérapeutique, la kinase mTOR. Cette dernière intervient dans la signalisation du stress cellulaire et son inhibition a de multiples conséquences fonctionnelles. La rapamycine est ainsi un immunosuppresseur puissant qui sert cliniquement à prévenir les rejets de greffe et à traiter un grand nombre de cancers. Cette molécule agit en se fixant à la protéine FKBP12 ; le complexe rapamycine-FKBP12 se fixant ensuite lui-même à la sous-unité FRB de la protéine mTOR. L’étude accomplie a permis de caractériser la façon dont se dissocie le complexe FRB-rapamycine-FKBP12 en déterminant l’orientation et la séparation relative des sous-unités au moment de leur rupture.
De façon plus générale cette pincette moléculaire s’apparente à un « détecteur universel » pouvant être utilisé pour caractériser de nombreuses autres associations moléculaires, soit dans le domaine du vivant (entre protéines ou entre protéine et médicament), soit dans le domaine de la chimie et des matériaux, (entre nanoparticules ou entre nanoparticule et petite molécule).
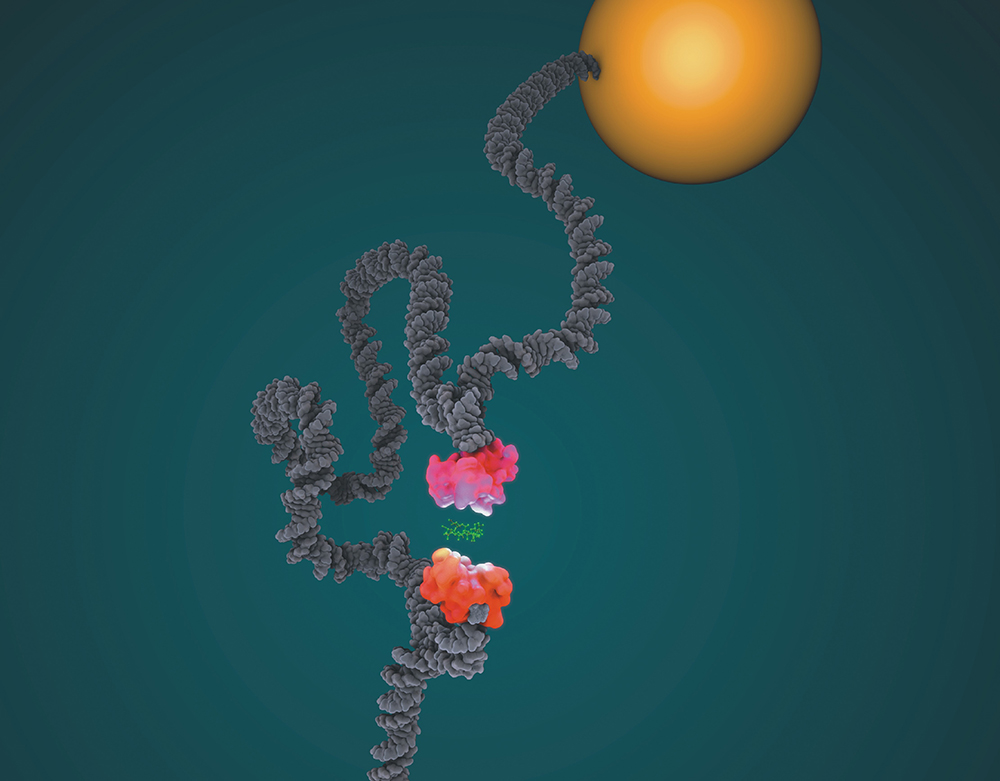
Figure : Une molécule d’ADN est transformée en pincette moléculaire par l’ajout d’un « pont » interne synthétique. L’ADN est ensuite fixé par une extrémité à une bille magnétique et par l’autre extrémité à une surface de verre. Des molécules d’intérêt sont greffées sur la pincette, et leur interaction est détectée en temps réel en observant l’état de la pincette (pont « ouvert » ou pont « fermé »).
Pour en savoir plus
A modular DNA scaffold to study protein-protein interactions at single-molecule resolution
Kostrz D, Wayment-Steele HK, Wang JL, Pande VS, Strick TR & Gosse C
Nature Nanotechnology 23 September 2019 doi: 10.1038/s41565-019-0542-7