Le mécanisme derrière la fidélité de la translocation du ribosome eucaryote
Dans cet article publié dans la revue Nature, les scientifiques décrivent pour la première fois le ribosome eucaryote piégé dans un stade précoce du processus de translocation, lorsque l'ARN messager couplé aux ARN de transfert est précisément transporté à travers le ribosome. En utilisant une technique de cristallographie à haute résolution, ils apportent des données sur le « chaînon manquant » à la compréhension actuelle de ce mécanisme de translocation, et sa précision en termes atomiques.
La traduction du code génétique en protéines s’effectue, au sein du ribosome, par des répétitions de mouvements synchrones d’un codon d’ARN messager (ARNm) et d’ARN de transfert (ARNt) à la fois. Ce cycle de mouvements, appelé translocation, est étroitement régulé car les erreurs dans le mouvement des codons d’ARNm appariés aux anticodons d’ARNt provoquent un décalage du cadre de lecture de l’ARNm qui peut conduire à la formation de protéines aberrantes ou tronquées qui, si elles ne sont pas éliminées, peuvent déclencher des effets cellulaires délétères. Chez les eucaryotes, le processus complexe de translocation est assuré par la translocase/facteur d’élongation 2 (eEF2) qui catalyse le processus et contribue activement à sa fidélité. Malgré d’énormes efforts, les connaissances des structures des complexe de translocation procaryote et surtout eucaryote restent très limitées en raison d’une résolution insuffisante et de l’hétérogénéité de composition des complexes.
Les scientifiques présentent la première structure cristallographique à haute résolution du ribosome eucaryote 80S provenant de Saccharomyces cerevisiae dans un contexte in vivo où le ribosome est piégé dans un stade précoce de la translocation. Cet état n’a jusqu’à présent jamais été décrit ni pour les ribosomes bactériens, ni pour les ribosomes eucaryotes. Les modèles montrent comment le centre de décodage du ribosome libère le duplexe codon-anticodon, permettant ainsi le mouvement de ces derniers au sein du ribosome. Les données permettent également de préciser la fonction de la translocase eEF2 en tant que « cliquet » qui, de plus, détermine la directionnalité de la translocation. Ils permettent de mettre en évidence le mécanisme par lequel les éléments spécifiques au ribosome eucaryote, ainsi que l’eEF2 et l’ARNt subissent les réarrangements moléculaires à grande échelle afin d’assurer le maintien de la fidélité du cadre de lecture de l’ARNm au cours de la translocation.
Les données obtenues permettent d’apporter le « chaînon manquant » à la compréhension actuelle du mécanisme de translocation et de sa fidélité au niveau atomique. Le mécanisme suggéré, mis à part son importance fondamentale, peut aider à la compréhension du décalage du cadre de lecture activement exploité par de nombreux virus, notamment le SARS-CoV-2 et le VIH pour leur recodage de messages génétiques.
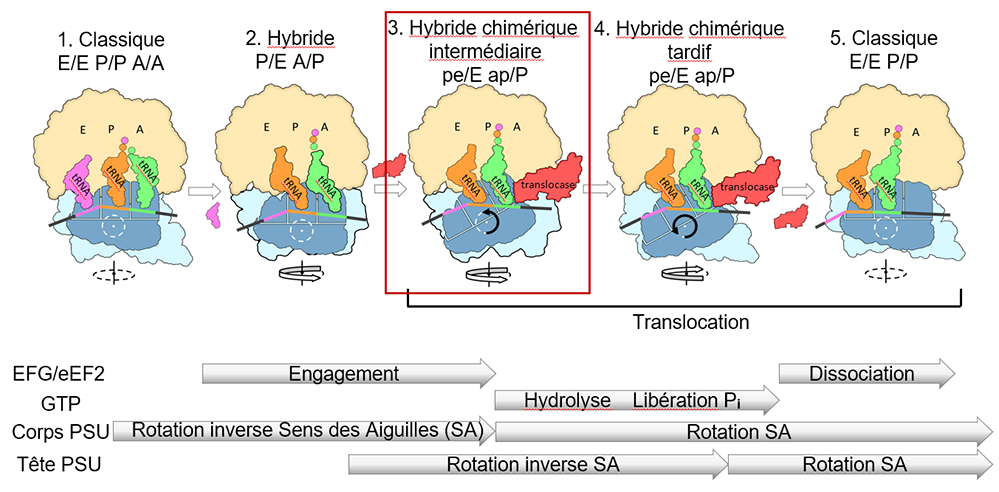
Figure : Intégration des études structurelles et cinétiques de la translocation. Le schéma détaille la translocation en se basant sur la structure cristalline du complexe de translocation intermédiaire précoce, rapportée ici dans le cadre en rouge, ainsi que sur les structures déjà publiées de translocation intermédiaire tardif obtenues grâce à la cryo-EM. Il met en lumière la séquence d’évènements qui compose la translocation dans les détails atomiques décrite dans l’article original. Le schéma montre notamment les rotations de la tête et du corps de la petite sous-unité du ribosome (PSU).
Pour en savoir plus :
Accuracy mechanism of eukaryotic ribosome translocation.
Djumagulov M, DemeshkinaN, JennerL, RozovA, YusupovM & Yusupova G
Nature 1er décembre 2021, DOI: 10.1038/s41586-021-04131-9.
Contact
laboratoire
Institut de génétique et de biologie moléculaire et cellulaire (IGBMC) (CNRS/Inserm/Université de Strasbourg)
1, rue Laurent Fries
67404 Illkirch Cedex